华人学者携手David Liu课题组!首次用腺嘌呤碱基编辑器成功治疗酪氨酸代谢病!
| 导读 | 遗传性I型酪胺酸血症(hereditarytyrosinemiatypeI,HT1)是一种肝脏遗传病,主要是由于延胡索酰乙酰乙酸水解酶(FAH)基因的一个A->G点突变导致(图2a)。HT1病人需要遵从严格的饮食要求和接受尼替西农(nitisinone, NTBC)的治疗。 |
遗传性I型酪胺酸血症(hereditary tyrosinemia type I, HT1)是一种肝脏遗传病,主要是由于延胡索酰乙酰乙酸水解酶( FAH)基因的一个A->G点突变导致(图2a)。HT1病人需要遵从严格的饮食要求和接受尼替西农(nitisinone, NTBC)的治疗。如果治疗失败,患者会出现肝功能衰竭和肝癌。2月25日,在《自然》子刊《Nature Medical Bioengineering》上刊登的一篇科学论文中,麻省大学的Wen Xue课题组、武汉大学的Hao Yin课题组和Broad 研究所David Liu课题组合作使用基于CRISPR系统的碱基编辑器,第一次成功地在成体小鼠模型中治疗了由于G->A基因突变导致的这种罕见肝脏疾病。Chun-Qing Song ( 宋春青,西湖大学研究员) 和Tingting Jiang 是本文的共同第一作者。
CRISPR-Cas9基因编辑系统由于其便捷、可编程的特点,自问世以来获得了生命科学研究领域的广泛关注。科学家可以利用CRISPR-Cas系统在基因组的特定位点对DNA序列进行编辑。可是CRISPR基因编辑系统的工作的过程中会产生双链DNA断裂,这导致编辑副产物的出现。
为了弥补这一缺陷,Broad 研究所华人学者David Liu实验室在CRISPR系统的基础上,开发出了碱基编辑器,从而实现了直接将一个碱基或碱基对转换成另一个碱基或碱基对(例如将A-T转换成G-C),并能够在不分裂的细胞中有效地实现点突变,而且基本不会产生过多的编辑副产物(图1)。这种碱基编辑器具有治疗单基因点突变造成的遗传病的极大潜力。
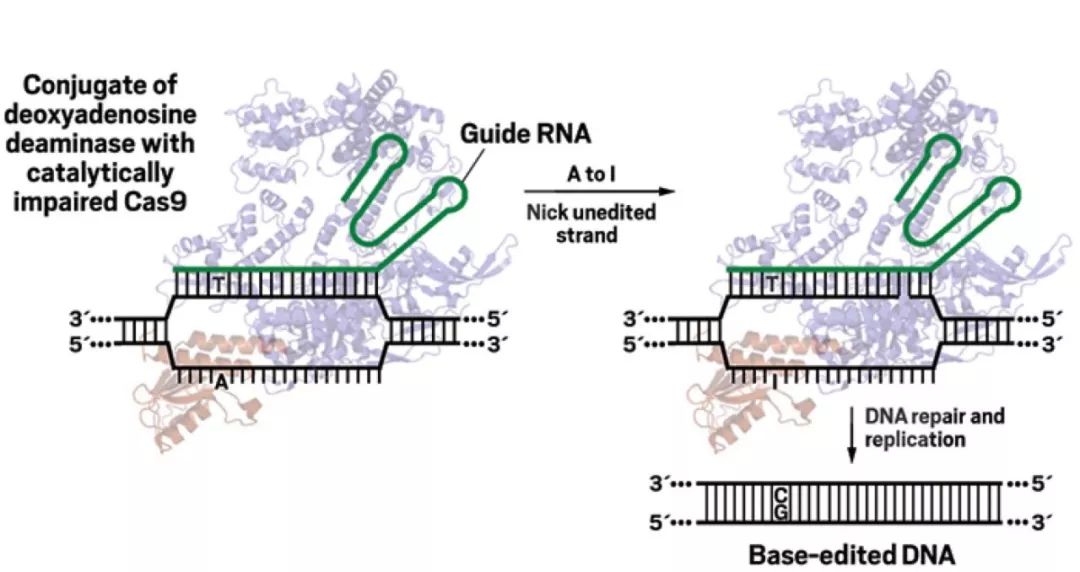
图1腺嘌呤碱基编辑器abe作用原理
在本研究中,研究人员将腺嘌呤碱基编辑器( adenine base editors, ABEs ) ABE6.3和CRISPR系统一起通过尾静脉高压注射的方式导入到患有遗传性I型酪胺酸血症(hereditary tyrosinemia type I, HT1)的成年老鼠肝脏内。
实验结果表明,接受ABE6.3碱基编辑疗法的HT1小鼠,肝脏中经过编辑的肝细胞数量稳定, 产生了大量含有正常功能的FAH蛋白(图2)。而且接受碱基编辑疗法的小鼠的肝脏功能和生存率大大提高。不仅如此,研究人员还对ABE6.3碱基编辑器进行了优化改进到升级版本RA6.3(图3)。细胞跟体内实验结果证实, 升级版碱基编辑器RA6.3的编辑效率约是初始版本ABE6.3的2倍。此研究表明了碱基编辑器在治疗成年老鼠体内肝脏遗传病的可行性。
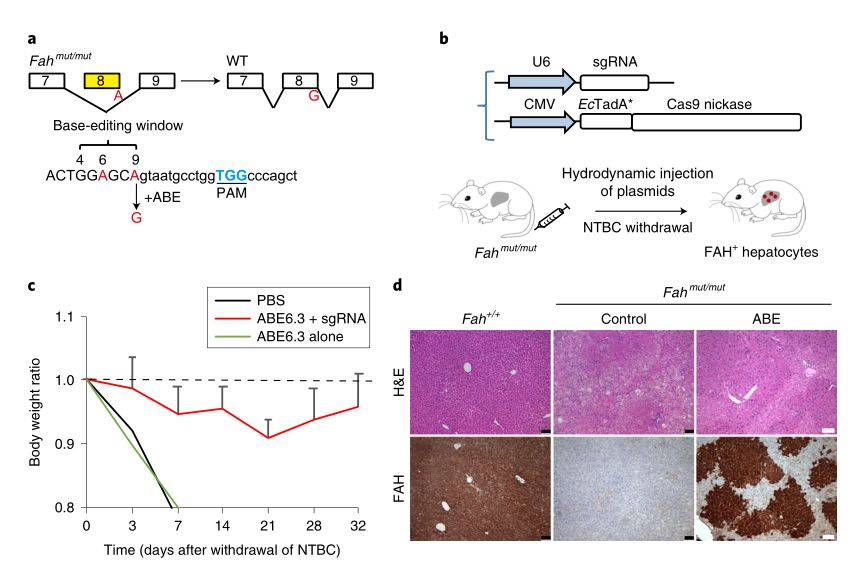
图2 abe修复了患有遗传性I型酪胺酸血症(ht1)的成年小鼠
a) HT1中, Fah基因外显子8最后一个碱基G->A的点突变导致所表达的蛋白外显子8缺失,进而使FAH蛋白丧失功能, ABE碱基编辑器通过诱导A->G进而修复FAH蛋白。
b) 将携带Fah基因的导向RNA (sgRNA)的质粒与 ABE质粒一起通过小鼠尾静脉导入到携带HT1疾病小鼠的肝脏中。
c) ABE跟sgRNA 的导入使HT1疾病小鼠维持在正常体重水平。
d) IHC结果显示 ABE跟sgRNA一起修复了小鼠的肝脏细胞。
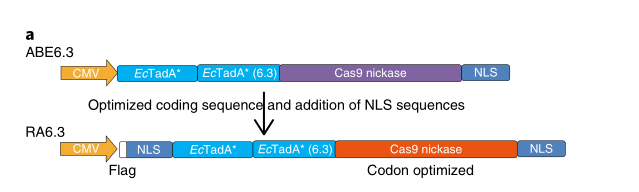
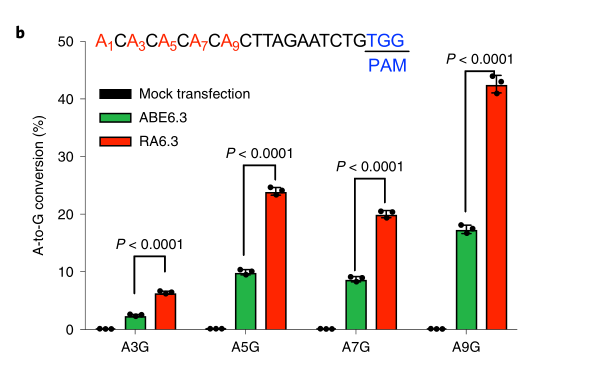
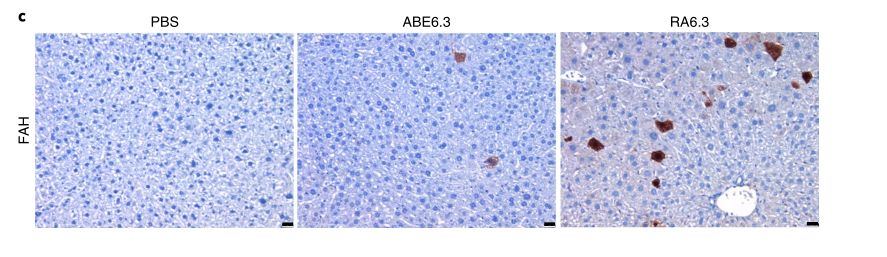
图3 碱基编辑器ABE6.3的升级版本ra6.3大大提高了碱基编辑效率
a) 通过优化ABE6.3中dCas9的编码序列和添加核定位序列(NLS)得到升级版 ABE6.3(RA6.3)。
b) 在293T细胞中的结果显示, 优化版RA6.3的编辑效率相对于原始ABE6.3大大提高。
c) 与ABE6.3相比,优化版RA6.3在HT1疾病小鼠中介导的肝细胞初始修复数目明显增多。
迄今为止,已知人类致病突变的最大类别是点突变(也称为单核苷酸多态性( SNP ) ),因此,碱基编辑器对于SNPs引起的遗传疾病的研究和治疗具有巨大的推动作用。
参考文献
Song, C.Q. et al. Adenine base editing in an adult mouse model of tyrosinemia. Nature Biomedical Engineering (2019).
Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature (2017).
Jon Cohen, Base editors’ open new way to fix mutations. Science (2017).
Rees, H.A. & Liu, D.R. Base editing: precision chemistry on the genome and transcriptome of living cells. Nature Reviews Genetics (2018).
Aponte, J.L. et al. Point mutations in the murine fumarylacetoacetate hydrolase gene: Animal models for the human genetic disorder hereditary tyrosinemia type 1. Proc Natl Acad Sci U S A (2001).
Song, C.Q. & Xue, W. CRISPR-Cas-related technologies in basic and translational liver research. Nat Rev Gastroenterol Hepatol (2018).
(转化医学网360zhyx.com)
 腾讯登录
腾讯登录






还没有人评论,赶快抢个沙发