【Nature子刊】多种癌症早发现!无细胞DNA的“无用之用”!
| 导读 | 早期发现各种癌症是癌症医学的重要部分,癌症在早期诊断和治疗时具有更好的预后和生存率。在大多数情况下,诊断检查是在癌症出现症状后才进行的,并且经常错过治疗时间。基于cfDNA的非侵入性癌症筛查可用于多种癌症早期检测和定位。 |
近日,国外一研究机构在《Nature: Communications》上发表了名为“Integrative modeling of tumor genomes and epigenomes for enhanced cancer diagnosis by cell-free DNA”的文章,研究人员对无细胞DNA(cfDNA)进行了全基因组测序,其生成的算法模型可在早期阶段提前检测出多种癌症。

https://www.nature.com/articles/s41467-023-37768-3
研究背景
01
无细胞DNA或游离DNA(Cell free DNA,cfDNA),是释放到血浆中降解的DNA片段,存在于人体的各种体液中,随组织损伤、癌症和炎症反应等发生浓度变化。
无细胞DNA (cfDNA)非侵入性筛查有很大希望发现多种癌症的早期阶段。循环肿瘤DNA (ctDNA)能反映出肿瘤特异性遗传和表观遗传改变。此外,ctDNA片段相较于正常的cfDNA片段而言要更短。利用ctDNA的特征,目前已研究出多种方法检测癌症。除了依赖于靶向方法,研究人员基于DNA甲基化模式以及基因组碎片模式新开发了一种全基因组方法。
研究方法
02
全基因组测序(WGS)在检测低负担疾病方面比靶向深度测序更精准。研究人员人认为,通过利用大量突变的累积信号,可以实现对微小残留疾病的超灵敏监测。然而,这种方法只能追踪患者肿瘤组织的初始突变特征,而不能从头识别突变。
到目前为止,还没有研究人员尝试基于cfDNA的全基因组测序用于从头癌症检测,这主要是因为变异调用和cfDNA过滤不准确。
与靶向深度测序不同,在没有正常对照的情况下,cfDNA的从头突变调用的可信度较低。为弥补这一点,研究人员利用来自大型健康队列的参考cfDNA测序数据过滤种系突变和各种类型的人为因素。
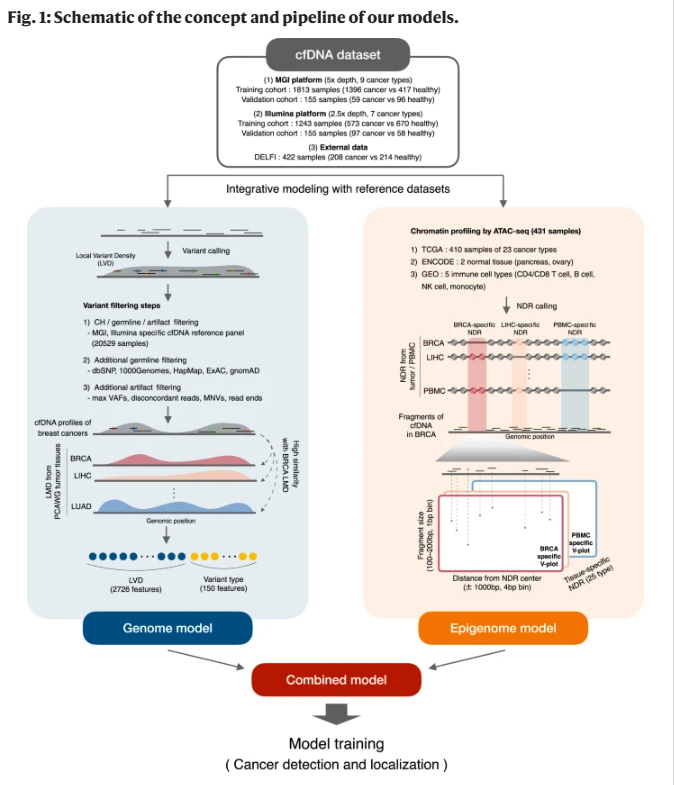
具体而言,就是尝试使用cfDNA全基因组测序进行多种癌症检测,该“基因组模型”集成了来自健康队列的大规模参考cfDNA数据以及PCAWG项目的肿瘤组织突变数据。在此过程中结合系统cfDNA变异过滤和大规模参考数据库时,基因组模型可以捕获肿瘤来源的突变。在工作中,研究人员使用cfDNA全基因组测序与“表观基因组模型”进行多癌症检测,该模型使用染色质开放性测序技术(ATAC-seq)集成了泛癌症全基因组染色质谱,还基于ATAC-seq数据为各种肿瘤类型的TCGA(癌症基因组数据集)样本构建了组织特异性NDR谱。
基因组模型采用了彻底的变异过滤过程(图1)。研究人员还对20,529个健康正常的cfDNA样本进行了低通量全基因组测序。该数据集作为正常的参考面板,用于过滤潜在的生物和技术噪声,并在公共数据库的基础上进行额外种系和人工制品筛选。该过程倾向于过滤掉来自健康样本的变体,同时保留来自患者样本的变体。
为测试算法,研究人员在两个不同的测序平台上为3366个样本生成了cfDNA WGS数据。以较大批次的样本作为训练队列,剩下的批次用于验证。除了基因组模型和表观基因组模型的个体性能外,还评估了两种模型联合使用的效果。
https://www.nature.com/articles/s41467-023-37768-3
研究意义
03
从头检测癌症,特别是在早期阶段,仍然具有挑战性。为充分利用全基因组cfDNA筛查的潜力,研究团队开发了一种分析策略,策略经过cfDNA和组织样本的大量训练和参考数据支持,结合了肿瘤基因组和表观基因组,显示出前所未有的癌症检测准确性。
在这项研究中,研究人员不仅确定了哪些特征在模型的开发和性能中发挥了重要作用,而且还从癌症的遗传和表观遗传特征方面研究了这些特征与肿瘤生物学的相关性。当结合基于肿瘤生物学的综合建模时,越来越多的cfDNA分析参考和训练数据有可能实现超灵敏的早期癌症检测和准确的癌症诊断。(转化医学网360zhyx.com)
参考资料:
https://www.nature.com/articles/s41467-023-37768-3
注:本文旨在介绍医学研究进展,不能作为治疗方案参考。如需获得健康指导,请至正规医院就诊。
 腾讯登录
腾讯登录







还没有人评论,赶快抢个沙发