Nature Medicine:肿瘤全基因组测序助力癌症个体化治疗
| 导读 | 近年来,随着全基因组测序所需时间和成本的降低,个体化治疗将造福更多的癌症患者。近日发表于《NatureMedicine》的一项研究表明,开发新的机器学习算法对肿瘤全基因组序列信息进行分析,有助于预测患者的预后,帮助患者选择最佳的治疗方案。 |

近年来,随着全基因组测序所需时间和成本的降低,个体化治疗将造福更多的癌症患者。近日发表于《Nature Medicine》的一项研究表明,开发新的机器学习算法对肿瘤全基因组序列信息进行分析,有助于预测患者的预后,帮助患者选择最佳的治疗方案。

全基因组测序(Whole-genome sequencing,WGS)技术可获取患者几乎全部的遗传信息,包括癌细胞和健康细胞的全基因组信息,进而可在单核苷酸、拷贝数、表观遗传修饰等层面发现癌细胞的异常。
为了挖掘WGS的临床价值,剑桥大学的研究人员与瑞典隆德大学研究人员合作开展了一项名为SCAN-B (Sweden Cancerome Analysis Network–Breast)的全人群项目(ClinicalTrials.gov ID:NCT02306096),该项目从2010年开始在瑞典招募乳腺癌患者,迄今已收集了大量的临床数据。
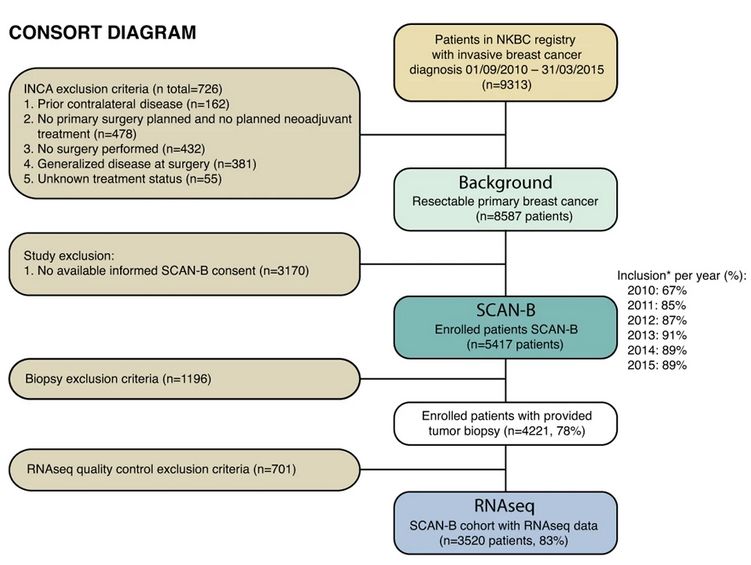
瑞典乳腺癌基因组分析网络(SCAN-B) 项目流程
在SCAN-B项目中,研究人员对2010至2015年间SCAN-B中的254名三阴性乳腺癌(triple-negative breast cancer, TNBC)进行了全基因组测序。三阴性乳腺癌是指雌激素受体、孕激素受体及人表皮生长因子受体2均阴性的乳腺癌患者,约占乳腺癌的9%,预后较差。
随后,研究人员应用了一种叫做“HRDetect”的机器学习算法对肿瘤进行了分类。最初开发此算法是为了检测具有BRCA1/BRCA2基因突变签名的肿瘤,BRCA1/BRCA2基因中的任一个发生突变都会大大提高乳腺癌患病风险。目前对于具有BRCA1/BRCA2基因突变的乳腺癌患者可以采用一种新的靶向性药物PARP抑制剂进行治疗。
根据HRDetect算法对三阴乳腺癌患者肿瘤基因组序列的评分,研究人员将患者分为高(HRDetect-high)、中(HRDetect-intermediate)、低(HRDetect-low)三类。59%的患者具有同源重组修复缺失(HRDetect-high):其中67%为BRCA1/BRCA2的生殖系/体细胞突变,BRCA1启动子高甲基化,RAD51C高甲基化或PALB2双等位基因丢失。
HRDetect算法提供了一种独特的诊断信息。研究人员发现,与得分低(HRDetect-low)的患者相比,得分高(HRDetect-high)的三阴性乳腺癌患者治疗效果最好,对PARP抑制剂也可能更加敏感。
令人惊讶的是,得分中等(HRDetect-intermediate)的患者治疗效果最差,即使一些患者肿瘤具有一些较好的药物靶点,预后依然很差。因此,对于得分不高的患者,有必要采取新的治疗方案。得分低(HRDetect-low)的患者预后也较差,然而,在这些患者肿瘤的WGS序列信息也表现异常,可能是由临床试验中使用固定一些药物导致,比如免疫检查点抑制剂(PD-1)或AKT抑制剂。
该研究第一作者Johan Staaf博士说:“三阴乳腺癌很难治疗,但通过全基因组测序,我们可以鉴别出哪些三阴性乳腺癌患者对当前临床上使用的药物比较敏感。重要的是,这种方法可以让我们为研究预后差相关机理提供了线索,并据此开发针对此类患者的新药。”
随着测序技术的飞快发展,全基因组测序已经可以在24小时内完成,而测序数据的分析可以在24-48小时内完成。因此,理论上,可以为每一位患者提供全基因组测序和分析,并依据患者自己的肿瘤基因组信息进行最优治疗方案的选择。
“全基因组测序为癌症个体化治疗开辟了新的道路,”共同第一作者Nik Zainal博士说,“过去,大量数据的管理和分析是其广泛应用的主要障碍。但是现在我们正缩短数据分析时间,让每一名患者可以得到个体化治疗,这将会在很大程度上改变癌症的治疗,甚至是一些难治癌症的治疗。”
参考文献:
Whole-genome sequencing of triple-negative breast cancers in a population-based clinical study, Nature Medicine (2019).
DOI: 10.1038/s41591-019-0582-4
推荐内容:
-
第二届上海国际癌症大会分论坛:肿瘤免疫与微环境
-
第二届上海国际癌症大会主论坛:肿瘤免疫专场
-
第二届上海国际癌症大会分论坛:肿瘤代谢与微环境
-
第二届上海国际癌症大会分论坛:肿瘤临床及转化
-
第二届上海国际癌症大会日程公布:三人同行一人免单活动即将截止!
(转化医学网360zhyx.com)
 腾讯登录
腾讯登录







还没有人评论,赶快抢个沙发