使用自适应采样的无 PCR 靶向测序对遗传性癌症进行全面分析
| 导读 |
仅需一张 MinIONTM 测序芯片,即可对人类基因组进行简单的靶向测序,在单一数据集下实现 SNP 识别和定相、甲基化分析,以及低通量全基因组拷贝数变异分析。
联系方式:publications@nanoporetech.com
更多信息请访问:www.nanoporetech.com 和 publications.nanopore... |
仅需一张 MinIONTM 测序芯片,即可对人类基因组进行简单的靶向测序,在单一数据集下实现 SNP 识别和定相、甲基化分析,以及低通量全基因组拷贝数变异分析。
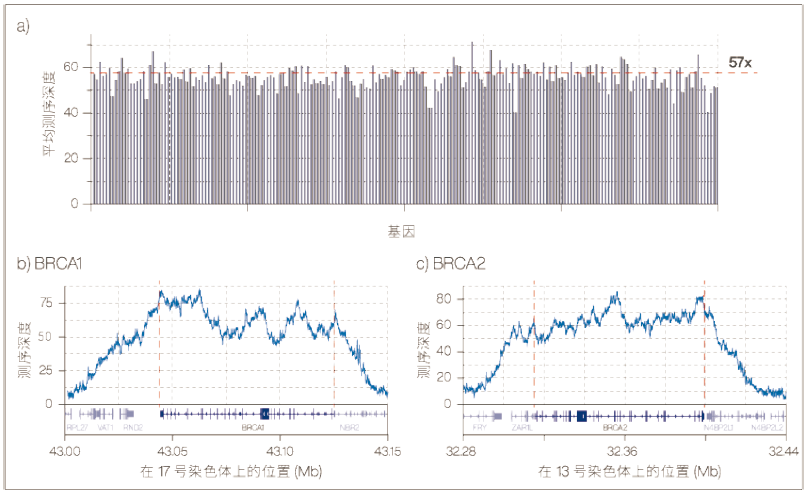
图1 自适应采样可覆盖的范围 a) >200 个癌症相关基因 b) BRCA1 c) BRCA2
自适应采样支持在测序运行期间靶向基因组区域
在自适应采样技术下,当 DNA 链穿过纳米孔时,可以对其进行碱基识别。如果读长序列的前 400-500 个核苷酸能与预设的靶向区域比对,则允许该读长序列通过。如果比对不成功,整个纳米孔的电势将逆转,从而拒绝 DNA 链通过。然后再开启纳米孔,以便后续模板分子通过。在此介绍我们设计的一个组合,可靶向整个基因组中的 201 个遗传性癌症相关基因和 >15,000 个启动子区域。经过 72 小时的测序,癌症基因的平均覆盖度为 57x(图 1a)。沿着极长靶基因(例如 BRCA1 和 BRCA2),从一端到另一端的覆盖度都相当均匀(图 1b 和 1c)。
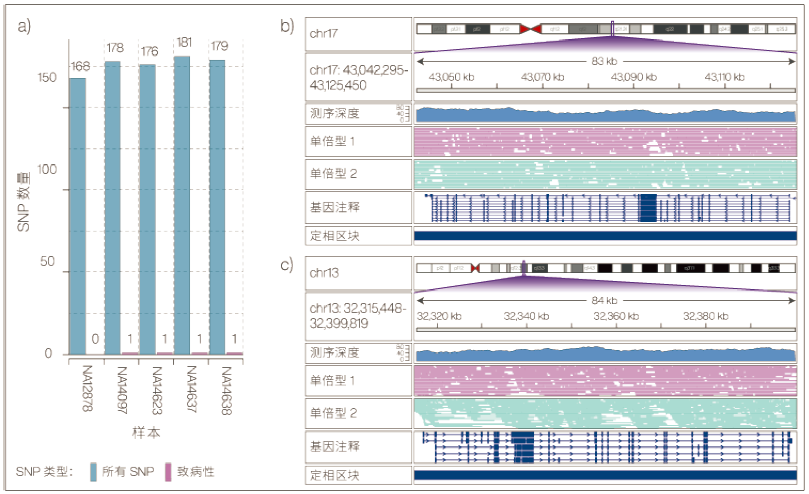
图2 SNP 识别 a) 乳腺癌和卵巢癌样本 b) BRCA1 c) BRCA2 中的定相
在癌症样本中富集的靶向区域进行 SNP 识别,以发现致病性变异
我们使用 clair3 识别 SNP,并将识别结果与 ClinVar 相交(图 2a)。我们在 NA14097 BRCA1 外显子 5 中发现了一个 T > G SNP(其导致一种错义突变,可产生非功能性蛋白),在 NA14637 BRCA1 外显子 13 中发现了一个无义突变,在 NA14638 的 BRCA1 内含子 5 中发现了一个隐匿剪接位点。尽管 NA14623 是 BRCA 阴性乳腺癌样本,但我们在 PALB2 中发现了致病性变异。PALB2 是形成 BRCA1-PALB2-BRAC2 修复蛋白复合物的关键蛋白。对这些复杂基因进行定相,可将它们的单倍型完全区分开来(图 2b 和 2c),这对化合物杂合性研究很重要。定相还可区分靶基因及其假基因。
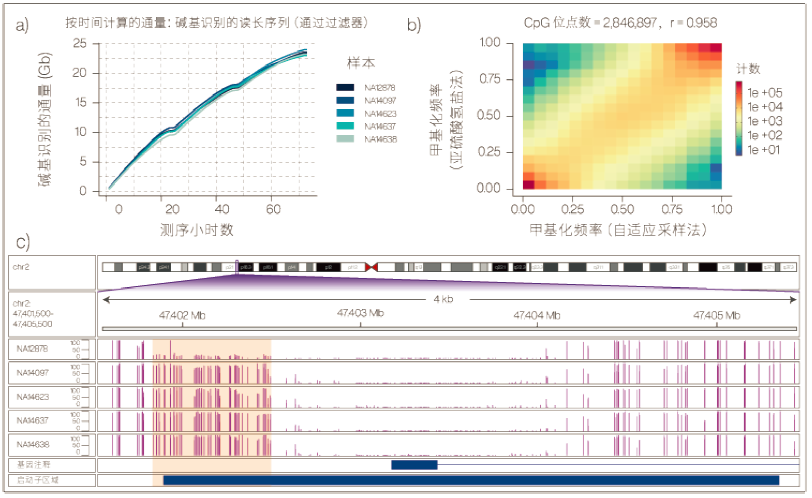
图3 自适应甲基化分析 a) 通量 b) 与亚硫酸氢盐法的相关性 c) MSH2 基因
采用自适应采样的靶向测序是无 PCR 的,因此保留了甲基化信号
我们还在自适应采样 bed 文件中纳入了超过 15,000 个启动子区域,这些启动子区域均含有 >4 个 CpG,因此可进行一种约化表示的甲基化识别。15,000 个启动子区域中,大多数的测序深度足以提供稳健的、以 CpG 为单位的甲基化识别(图 3a)。将自适应采样的甲基化识别结果与同一样本的亚硫酸氢盐数据集进行比较,发现在 280 万个 CpG 位点上,两种方法的相关系数为 0.958(图 3b)。在五个基因组中,许多位点显示出迥异的甲基化模式。比较所有癌症样本和 NA12878 非癌症对照,发现在已知错配修复基因 MSH2 的启动子/增强子区域,高甲基化存在明显差异(图 3c)。

图4 全基因组 CNV 分析 a) 测序深度 b) PIP 基因中的缺失
使用被拒绝的脱靶读长序列分析拷贝数变异

 腾讯登录
腾讯登录



还没有人评论,赶快抢个沙发