异质性批量肿瘤样本中的克隆和单倍型特异性拷贝数变异推断
| 导读 | 通过全基因组纳米孔测序,表征异质性肿瘤中的单倍型特异性大规模拷贝数变异(CNV)和新型低/高甲基化。 |
通过全基因组纳米孔测序,表征异质性肿瘤中的单倍型特异性大规模拷贝数变异(CNV)和新型低/高甲基化。
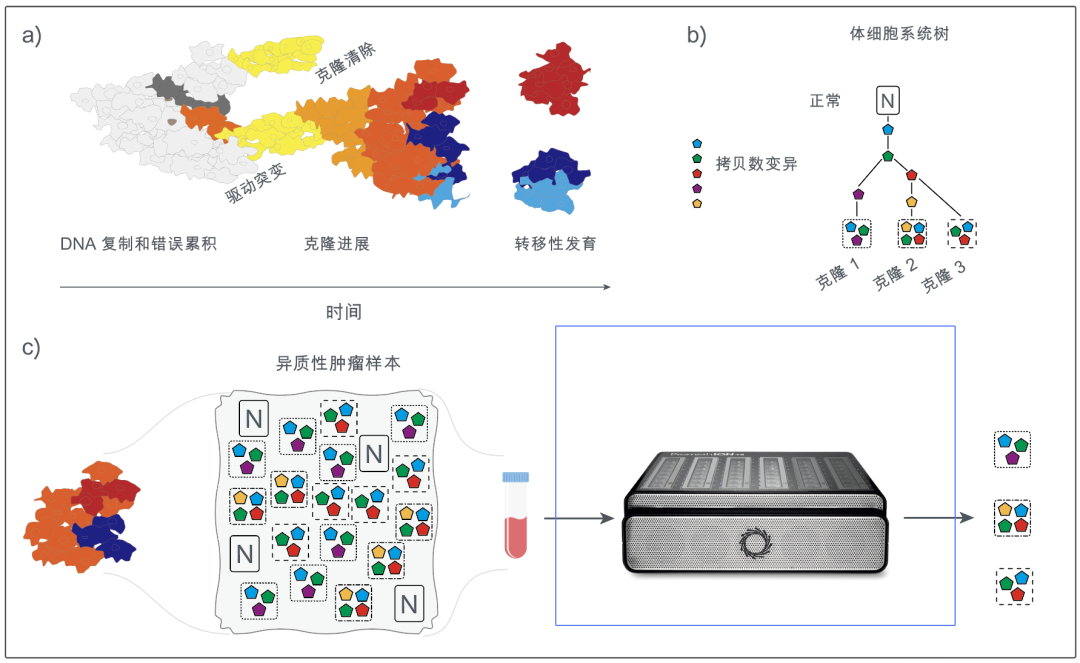
图1 癌细胞克隆进展和体细胞突变累积的图示
癌症疾病很复杂,可视为一种体细胞进化过程,伴随克隆细胞增殖和克隆清除(驱动因素是有益于生存/逃逸的基因组突变发生累积),同时选择性压力不断变化(图1a)。在癌症进程中,大量体细胞基因组CNV会沿着体细胞系统树的分支累积。所得肿瘤由具有不同CNV谱的克隆组成(图1b)。CNV可扩增或删除基因组片段、染色体臂甚至整个染色体的一个或两个种系等位基因。通过全基因组测序(图1c)解析和量化肿瘤内异质性,可以为癌症治疗提供有用信息。
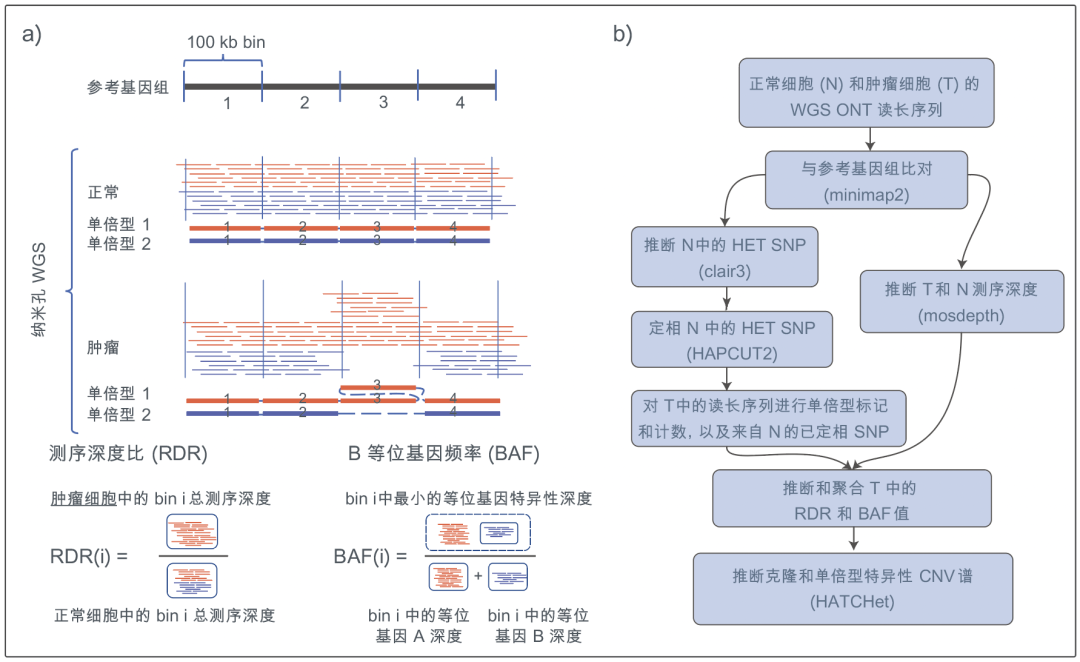
对bin片段参考基因组进行等位基因特异性CNV检测,其中主要信号为(1)给定 bin处的观察和预期测序深度之间的测序深度比(RDR),以及(2)给定bin的两个等位基因之一的读长序列占bin比对读长序列总数的B等位基因频率(BAF)(图2a)。对于匹配的正常细胞样本和一份或多份混合肿瘤细胞样本,我们的工作流程预计都可获得>30x的全基因组测序(WGS)读长序列。通过对单倍型标记的肿瘤读长序列计数,可获得肿瘤样本中的RDR和BAF值(在匹配的正常细胞样本中推断和定相HET SNP)。然后将RDR和BAF肿瘤值用作HATCHet方法的输入,得出所需的CNV谱(图2b)。
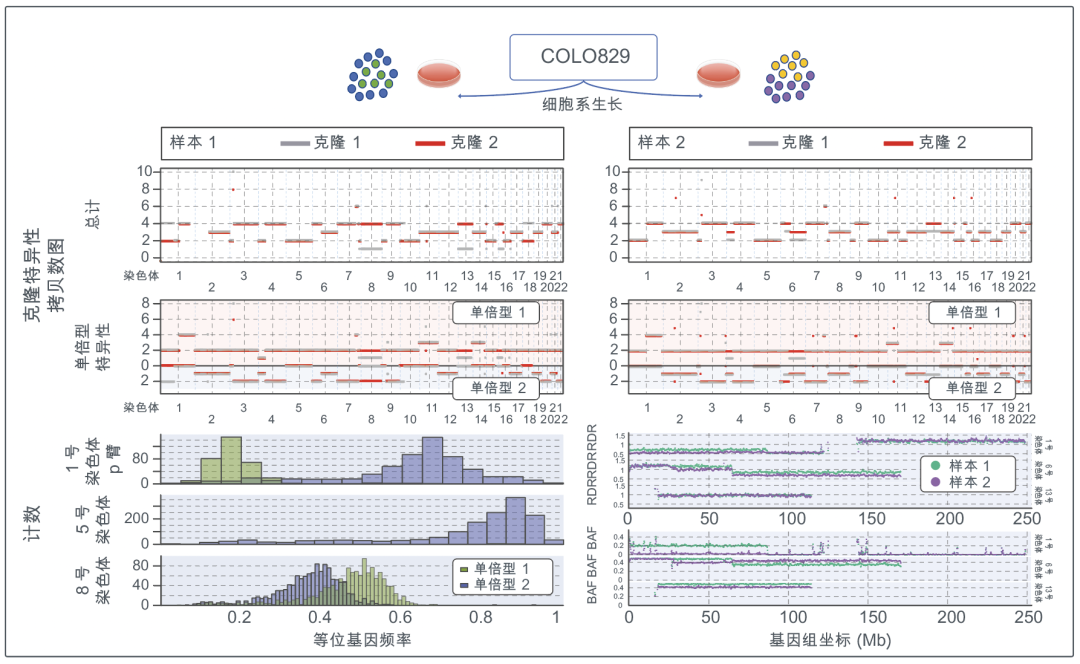
我们研究了COLO829(BL) 肿瘤(或正常)黑色素瘤细胞系。根据先前文献描述,这种细胞系具有高度非整倍体和细胞系生长异质性。我们研究了两份细胞生长平行样:使用我们的连接测序试剂盒,对从中提取DNA进行文库制备,然后在PromethIONTM平台上测序,达到>100x覆盖度。在两份平行样中,我们均获得一个2克隆描述,所得CNV和克隆分数得到了以下证据的支持:先前发表的COLO829表征结果,以及整个异质性CNV区域 HET SNP和RDR/BAF值的等位基因频率。我们观察到,在较低测序覆盖度水平下,克隆和单倍型特异性CNV推断结果具有稳定性,降至30x 肿瘤覆盖度时,CNV仍保持一致性(图3)。

图4 在 COLO829中观察到单倍型特异性甲基化

 腾讯登录
腾讯登录



还没有人评论,赶快抢个沙发