引领风向标,上海科技大学黄行许连发Nature Biot、Cell Res、Cell Dis,在基因编辑领域取得系列进展
| 导读 | Cell Research:上海科大与第三军医大强强联合,开发新型基因编辑技术 |
2018年7月17号,中国医学科学院北京协和医学院实验动物研究所张连峰课题组与上海科技大学黄行许课题组合作在Cell Discovery上在线发表了题“Highly efficient and precise base editing by engineered dCas9-guide tRNA adenosine deaminase in rats”的研究论文。该报道了这种以中国新型的腺嘌呤碱编辑器在大鼠碱基编辑中的应用。研究表明,该系统是一种可靠的大鼠基因组编辑工具,与传统的CRISPR / Cas9系统相比,该系统以更安全的方式诱导碱基转化。同时,证明了该系统在纠正人类遗传疾病相关突变方面的潜力。这也是黄行许课题组继2018年6月6号在Cell Research和2018年3月19号在Nature Biotechnology发表的相关文章后的又一重大突破。
1、Cell Research:上海科大与第三军医大强强联合,开发新型基因编辑技术((点击阅读)

2018年6月6号,上海科技大学黄行许课题组和第三军医大学陈洁平课题组合作在Cell Research上在线发表了题为“BE-PLUS: a new base editing tool with broadened editing window and enhanced fidelity”的研究论文。在这里,研究人员开发了一种新的编辑技术,通过将10个拷贝的GCN4肽融合到nCas9(D10A)中,将scFv-APOBEC-UGI-GB1引入到编码更大的C到U(T)范围(BE-PLUS) 。 新系统通过扩大窗口实现基础编辑,从而提高了基因组靶向范围。 有趣的是,新系统产生了更少的不必要的输入和非C到T转换。研究人员还通过诱导终止密码子(STOP),展示了它在整个基因组中的潜在应用。综合起来,BE-PLUS系统提供了一个新的编辑工具,增加了编辑窗口,提高了保真度。
由于CRISPR技术的效率和简便性,CRISPR技术被广泛应用于基因组操作中。在与靶序列结合后,Cas9引入DNA双链断裂(DSB),通过非同源末端连接(NHEJ)或同源 - 定向修复(HDR)途径,最终导致DSB站点的插入和缺失(插入)的积累。为了限制编辑过程中的DNA损伤,已经开发了CRISPRbased编辑策略,其需要直接靶向核苷酸转换而不形成DSB。 BE1是基础编辑器的原始版本,由催化失活形式的Cas9(dCas9)和rAPOBEC1组成。
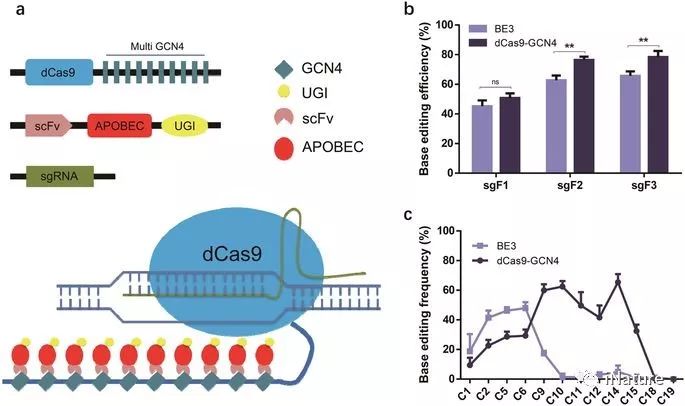
新的基编辑系统的生成,该系统扩展了编辑的窗口
BE2是最新版本,在dCas9的N末端含有一个尿嘧啶糖基化酶抑制剂(UGI),用于减少尿嘧啶N-糖基化酶(UNG)介导的碱基切除修复(BER)。BE2进一步优化至BE3,取代了dCas9由Cas9 nickase nCas9(D10A)提供。 BE3使得在非互补链DNA的4-8位(从PAM远端数起)处的胞嘧啶(C)直接和不可逆地转化为尿嘧啶(U)或胸腺嘧啶(T),并且诱导少得多的不想要的插入或易位。该方法已被用于在细胞,植物和动物中引入单核苷酸多态性,并能够在人类胚胎中操纵致病基因。最近,BE3也有被用于通过诱导终止密码子(iSTOP)来破坏基因。、
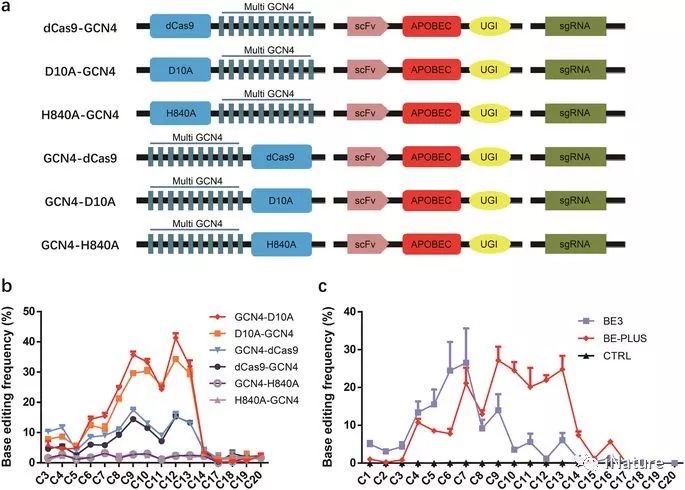
scFv-APOBEC系统的优化
尽管显示出巨大的潜力,但BE3可能会引入不需要的indels和意外的非C到T转换。此外,狭窄的编辑窗口限制了BE3的定位。已经成功应用了几种策略来提高BE3的保真度。例如,使用高保真nCas9(HF-nCas9)显着减少BE3.12的脱靶诱变。共同表达的游离尿嘧啶DNA糖基化酶抑制剂(UGI)与BE3增强了碱基编辑的效率和保真度。一种新的具有较长的rAPOBEC1和nCas9(D10A)接头和两个UGI复制子以减少尿嘧啶N-糖基化酶(UNG)介导的碱基切除修复(BER)的基因编辑器BE4在基础编辑中显示出更好的效率。此外,DNA末端结合Gam蛋白也被用于减少BE4引入不需要的插入缺失。同时,为扩大基因组靶向范围,已使用具有替代PAM序列的修饰Cas9变体。尽管如此,目前在整个基因组中进行的碱基编辑受限于sgRNA的第4至第8个可靶向位点。预计新战略将增加瞄准范围并提高编辑的保真度。
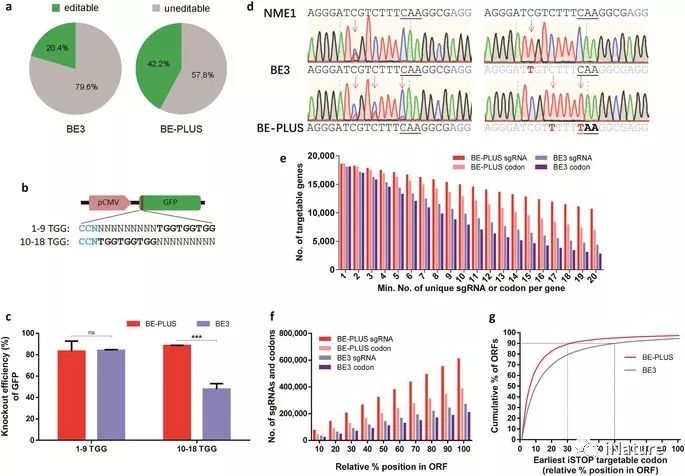
BE-PLUS在诱导终止密码子方面更有效
包含胞苷脱氨酶和催化缺陷的Cas9的基本编辑器(BE)已被广泛用于碱基编辑。 但是,BE的窄的编辑窗口限制了它的实用性。 在这里,研究人员开发了一种新的编辑技术,通过将10个拷贝的GCN4肽融合到nCas9(D10A)中,将scFv-APOBEC-UGI-GB1引入到编码更大的C到U(T)范围(BE-PLUS) 。 新系统通过扩大窗口实现基础编辑,从而提高了基因组靶向范围。 有趣的是,新系统产生了更少的不必要的输入和非C到T转换。我们还通过诱导终止密码子(STOP),展示了它在整个基因组中的潜在应用。综合起来,BE-PLUS系统提供了一个新的编辑工具,增加了编辑窗口,提高了保真度。
2、6篇Nature Biotec同时阐述CRISPR的最新进展(上科大贡献1篇,系统解析,值得收藏)(点击阅读)

各种物种中,通过将 apolipoprotein B mRNA编辑酶,催化多肽样(APOBEC)或激活诱导的脱氨酶(AID)胞苷脱氨酶家族成员与CRISPR-Cas系统组合开发的碱基编辑已经用于靶向C对T碱基编辑。然而,使用Cas9切口酶(nCas9)作为目前最活跃的BE中脱氨酶融合伴侣增加了不需要的插入和缺失(插入),非C-T碱基置换的频率,限制编辑富含G / C的PAM序列的区域。
Cpf1(Cas12a)是另一种Cas蛋白,它与Cas9的不同之处在于:Cpf1需要富含T的PAM序列(TTTV)用于目标DNA的识别; Cpf1(CRISPR RNA(crRNA))的sgRNA比Cas9短;并且Cpf1切割位点相对于间隔区DNA中的PAM序列位于远端和下游,而不是与Cas9的近端和上游。与Cas9相比,Cpf1也诱导更少的脱靶(OT)切割基因组。
为了解决以上问题,上海科技大学陈佳研究组及黄行许研究组及上海生科院杨力研究组合作通过将大鼠胞嘧啶脱氨酶APOBEC1融合到无催化活性的Lachnospiraceae细菌Cpf1上,开发了基于CRISPR-Cpf1的碱基编辑工具。 碱基编辑器可识别富含T的PAM序列,并催化人类细胞中的C至T转换,同时诱导低水平的插入缺失,非C至T替换和脱靶编辑。
该研究首次开发了一系列基于CRISPR-Cpf1的碱基编辑工具,它们能够以非常低的indel形式和非C-to-T替换进行有针对性的基础编辑,并且可以在富含A / T的区域进行编辑。 未来,预计其他Cpf1酶(例如,识别TTN PAM8的FnCpf1)或工程化的Cpf1 可用于进一步增强dCpf1的碱基编辑作用。
3、通过工程化dCas9-guide tRNA腺苷脱氨酶在大鼠中进行高效和精确的碱基编辑

大鼠是了解糖尿病、高血压和神经紊乱等人类疾病机制的参考实验动物模型。CRISPR/Cas9系统已被证明是一种高效、灵活的生成基因修饰鼠的工具。为减少副作用并扩大这一系统的应用作出了巨大努力。胞嘧啶碱基编辑器(CBE)含有CRISPR/Cas9基因工程的胞嘧啶脱氨酶,可以在不诱导双链DNA断裂或模板的情况下对哺乳动物基因组DNA进行修饰。CBE通过诱导C·G向T·A转换,其靶序列约有5个核苷酸窗口。
最近,同一组人开发了一种新的dCas9引导的tRNA腺苷脱氨酶,该酶能诱导A·T向G·C转换。与CBE一起,ABE能够在目标基因组序列12中引入所有四个核苷酸转变(C到T,A到G,T到C,G到A)。与野生型CRISPR/Cas9系统相比,这些基础编辑工具为人类疾病的基因校正提供了一种更为安全的方法。本文报道了这种新开发的腺嘌呤碱编辑器在大鼠碱基编辑中的应用。
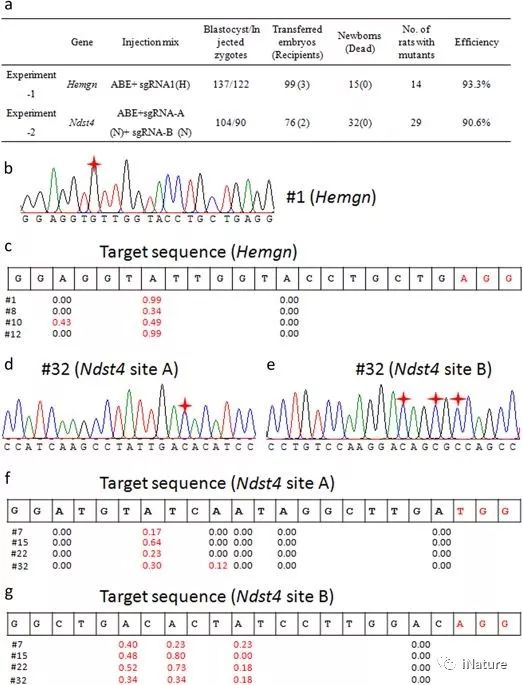
使用ABE系统对大鼠进行高效的碱基编辑
在这里,研究人员检查了4个样本(#1、#8、#10和#12)和NDST 4 sgRNA的14个非目标位点(未包括在内)的11个潜在非目标位点(未包括目标位点),以及4个样本(#7、#15、#22和#32)中NDST 4 sgRNA的14个非目标位点(不包括目标位点),这些样本使用Cas-OFFinder确定。对所有PCR产物进行测序和分析。在这些样本中没有发现非目标突变。为了进一步分析非目标效应,研究人员对选定的潜在非目标位点进行了目标深度测序。如上文所述,每个非目标站点的读取次数超过3M。正如预期的那样,没有检测到任何非目标效应。
接下来,对样本H-8(Hemgn靶向鼠)、H-10(Hemgn靶向大鼠)、N-15(NDST4-A和-B靶向大鼠)和N-32(NDST4-A和-B靶向大鼠)进行了全基因组测序(WGS),测序深度为×30。在Cas-OFFinder的基础上,获得了33718个HAMN-SgRNA的潜在离靶位点,20774个Ndst4sgRNA-A的潜在离靶位点,22547个NDST 4 sgRNA-B的潜在离靶位点,其中种子区的失配高达2-bp,NRG PAM在非种子区的失配达8-bp。在这些潜在的非目标位点中,只有几个潜在的非目标位点是从WGS数据中获得的。研究,进一步对这些潜在的非目标站点进行了排序,没有获得真正的非目标站点。结果表明,ABE系统是一种可靠的大鼠基因组编辑工具.
原文链接:
https://www.nature.com/articles/s41421-018-0047-9


个人简介:
1986年本科毕业于南昌江西农业大学牧医系;1995年在华南农业大学获硕士学位;1998年毕业于南方医科大学,获博士学位;1998年至2000年在中科院生物物理研究所从事博士后研究。2001年至2007年,在美国贝勒医学院从事博士后研究。2008年至2014年,受聘于南京大学模式动物研究所任教授、博导,2015年加盟上海科技大学生命科学与技术学院先后任终身副教授、研究员,2018年晋升为正教授。
研究方向:
在配子的发生过程中不仅要保证遗传和表观遗传的稳定,而且伴随发生一系列基本的生物学事件,如重编程和定向分化。配子发生提供了重编程和定向分化的理想模型。通过对配子发生的整体水平的研究,人们证实了重编程和定向分化是由转录因子和表观修饰调控的。当前生物学的研究前沿之一就是对这些重编程和定向分化过程中的转录因子和表观修饰调控机制及其生物学意义进行深入探讨。但由于技术的限制,很难进行位点特异的转录和表观修饰操作,阻碍了对配子发生过程中的重编程和定向分化的转录和表观遗传修饰调节的认识和理解。我们课题组的研究目的就是:研发特异性调节转录因子功能性反应元件以及表观遗传修饰的策略,并以此揭示配子发生过程中的转录和表观遗传修饰调节的机制及其生物学意义。(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发