麦肯锡:发布2025年12大颠覆技术 包括下一代基因组学和3D...
| 导读 | 麦肯锡发布了一项报告,罗列了将对2025年经济产生重大影响的12大颠覆技术,其中下一代基因组学列于第7,和3D打印技术(位于第9位的)同属于生物医药领域。
以下就是麦肯锡列举的颠覆性技术及其潜在的经济影响程度(含消费者盈余在内,即消费者并未支付的因创新而获得的价值),当然,这种影响评估只是粗略的,不会像 GDP 数字那么具体。
到 2025... |
麦肯锡发布了一项报告,罗列了将对2025年经济产生重大影响的12大颠覆技术,其中下一代基因组学列于第7,和3D打印技术(位于第9位的)同属于生物医药领域。
以下就是麦肯锡列举的颠覆性技术及其潜在的经济影响程度(含消费者盈余在内,即消费者并未支付的因创新而获得的价值),当然,这种影响评估只是粗略的,不会像 GDP 数字那么具体。
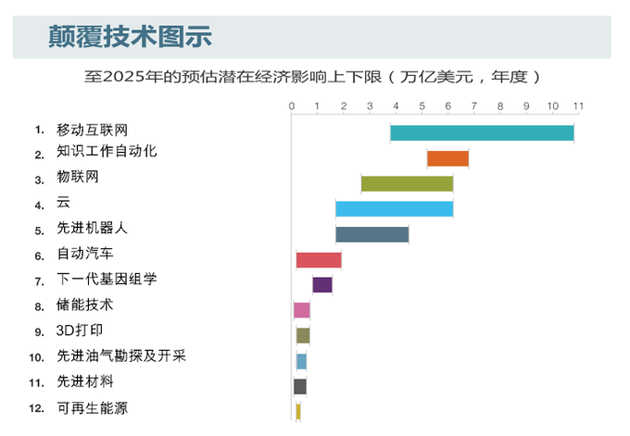
到 2025 年的影响力:
经济:0.7—1.6 万亿美元
生活:通过快速疾病诊断、新药物等延长及改善 75% 的生命
主要技术包括:先进 DNA 序列技术 ;DNA 综合技术;大数据及先进分析。
下一代基因组学中,尤为重要的是下一代测序技术、基因诊断以及基于遗传数据的个体化医疗。其中,下一代测序技术将产生高通量遗传数据,研究人员将这些海量的遗传数据与疾病进行关联(GWAS分析),从而提供疾病诊断和个体化医疗的靶向分子。
下一代基因组测序技术的3大产品
下一代基因组测序以单分子实时为特点,在纳米孔介质的基础上通过记录PH值、荧光和电流的改变,进而测出特定片段的碱基序列。目前,上市和待上市的下一代测序仪从技术特点上分为3个,它们是Life Technologies公司的Ion Torrent、Pacific Bioscience公司的PacBio RS和Oxford Nanopore Technologies公司的纳米孔测序仪。
Life Technologies公司的Ion Torrent
其核心技术是使用半导体技术在化学和数字信息之间建立直接的联系。在半导体芯片的微孔中固定DNA链,随后依次掺入ACGT。随着每个碱基的掺入,释放出氢离子,在它们穿过每个孔底部时能被检测到,通过对H+ 的检测,实时判读碱基。
Ion Torrent 半导体测序芯片的每个微孔里微球表面含有大约100 万个DNA 分子拷贝。测序时一个个核苷酸分子连续流过芯片微孔。如果核苷酸与特定微孔中的DNA 分子互补,则该核苷酸被合成到DNA 分子中,并且释放氢离子,该孔溶液的PH 发生变化。离子传感器检测到PH 变化后,即刻便从化学信息转变为数字电子信息。如果DNA 链含有两个相同的碱基,则记录电压信号是双倍的。
如果碱基不匹配,则无氢离子释放,也就没有电压信号的变化。这种方法属于直接检测DNA 的合成,因少了CCD 扫描,荧光激发等环节,几秒钟就可检测合成插入的碱基,大大缩短了运行时间。
Pacific Bioscience公司的PacBio RS
该测序仪的工作原理是,将基因组的DNA断开成许多很小的片段,这些小DNA片段制成溶液后滴入测序仪内的金属薄片上,金属薄片中分布着3000个纳米级小孔,这些纳米孔的直径不到70纳米,被滴入薄片上后,小DNA片段会分散到不同的纳米孔中。每个纳米孔中涂有一种特殊的酶——DNA聚合酶,DNA聚合酶 的特性是,能够沿着DNA片段的双链结构游动,在游动的过程中将DNA片段的双链结构打开,分成两个片段,也可为某个DNA单链找到对应的片段,重新组合 在一起,科学家将DNA聚合酶的作用形象地比喻成衣服的拉链,可开可合。
测序仪就是利用DNA聚合酶来“窃取人类的自然本性”的。一旦 DNA片段在纳米孔中分散完毕,向聚合酶分子滴入经过特殊处理的核苷酸溶液,谜底就会被揭开。核苷酸溶液中的每个核苷酸都用磷光染色剂做过标记,金属薄片 的底部也有一个缩微版光谱仪,一旦DNA片段中有核苷酸与之配对,标记物就会发出特定颜色的光,缩微版光谱仪就能检测并记录下这些闪光,测序仪从而记录下 每个DNA片段中的碱基对顺序。当闪光记录完之后,结果会输入计算机中,计算机破译出所有的DNA片段结果,并重新“组装”,让基因组恢复原样。
Oxford Nanopore Technologies公司的纳米孔测序仪
该测序仪有望在2013年上市,其工作原理是,单条DNA链将穿过蛋白孔,而第二种蛋白质将控制穿过的速度。随着碱基穿过纳米孔,它们改变了电流,带来特定的碱基组合。在理想的情况下,人们会看到每个碱基的典型模式,但目前的仪器将产生碱基三联体特有的模式,还需要通过计算机去卷积。
Oxford Nanopore Technologies公司在2012基因组生物学技术进展年会(AGBT)上宣布推出第一款商业化的纳米孔测序仪,引起了科学界的极大关注。纳米孔测序的读长很长,大约在几十kb,甚至100 kb;错误率目前介于1%至4%,且是随机错误,而不是聚集在读取的两端;数据可实时读取;通量很高(GridION有望在一天内以30倍覆盖度测序人类基因组);起始DNA在测序过程中不被破坏;以及样品制备简单又便宜。
具有突破意义的基因诊断
在最近召开的美国癌症研究学会(AACR)上,Mizuho Securities机构的分析师指出,基因诊断明显是肿瘤学的重要组成部分,即便大多数与会人士认为短期内分子诊断不能取代现存的诊断法,但是肿瘤基因组测序这一领域仍让多数人持乐观态度。报告提到了,更好的生物标记分子和诊断法被迫切需要。
研究人员和临床医生利用测序技术,以及基因组学、蛋白质组学和代谢组学方法去识别生物标记分子,促使其应用于遗传诊断、药物研发和临床药物测试的患者分类。
目前在分子诊断领域,最吸引眼球的新闻莫过于好莱坞女星兼导演安吉丽娜•朱莉因携带PCA缺陷基因而切除乳腺。她向《纽约时报》透露,因携带了母亲(因癌症去世)的癌症高危基因,所以不得不接受双乳乳腺切除预防乳癌手术治疗。这一PCA1缺陷基因让她患上乳癌几率高达87%,患卵巢癌几率50%。因此,朱莉2月主动选择做双乳乳腺切除降低患癌风险,手术已经在4月27日全部完成。
这一事件让“个人基因诊断”成为热门词汇。复旦大学生命科学院遗传学研究所副教授朱焕章接受采访时称:“基因检测的科学性是确定的,这也是未来的一个发展方向,将主要往个体化治疗、个体化诊断方向发展。每个人的情况都不一样,根据基因检测出个体的问题,就能有针对性地从根本上找出解决方案。”
个体化医疗前景值得期待
基因组时代已经到来,从基因组学角度实现传统医学的转化,通过分析基因组便可根据遗传信息掌握以前被称为“体质”的、由基因决定的个体特点,从而让个体化医学用于疾病预防和治疗的不同阶段。
美国贝勒人类基因组测序中心和有关机构联合发布一份报告称,贝勒大学医学院的研究人员及专家通过对一对双胞胎和他们的哥哥、父母的全基因组测序并进行对比分析,找到了引起这对双胞胎患有多巴反应性肌张力障碍疾病的突变基因,从而使医生能够对他们的病症进行针对性的治疗,并取得了良好的效果。贝勒人类基因组测序中心主任Richard Gibbs博士说:“我们有能力利用全基因组测序造福患者,这是一个巨大的进步。”
http://v.youku.com/v_show/id_XMjc2MjgwNzM2.html
个体化治疗患有多巴反应性肌张力障碍的双胞胎
另一个将基因组测序应用于个体化医疗的幸运儿是,患有白血病的华盛顿大学研究癌症的博士卢卡斯•沃特曼罹,在他病情恶化时,华盛顿大学里26台基因测序仪器中的一台,以及一台超级计算机,为沃特曼昼夜不停地运行。奇迹真的发生了!研究者们找到了元凶:一个正常基因FLT3表达 过于活跃,刺激了沃特曼癌细胞的快速生长增殖。更令人欣慰的是,他们找到了一种可能抑制问题基因表达的新药,这种新药之前只被批准用于治疗晚期肾癌。沃特曼成了第一个用这种药治疗白血病的病人。
基因组测序在个体化医疗成功运用的案例还是少数,已故的苹果公司前掌门人乔布斯当初对胰腺癌束手无策时,就曾联系医生进行过基因组测序和分析,不惜花费10万美元希望通过基因组测序找到胰岛细胞瘤的致病基因,从而实现个体化医疗。据乔布斯传记的作者艾萨克森透露,乔布斯本人也曾对此抱以巨大期望,他称自己要么是最早击败癌症的人之一,要么属于最后一批死于癌症的人。
文献链接:
The use of next-generation sequencing in clinical diagnosis of familial hypercholesterolemia.
Purpose:Familial hypercholesterolemia is a common Mendelian disorder associated with early-onset coronary heart disease that can be treated by cholesterol-lowering drugs. The majority of cases in the United Kingdom are currently without a molecular diagnosis, which is partly due to the cost and time associated with standard screening techniques. The main purpose of this study was to test the sensitivity and specificity of two next-generation sequencing protocols for genetic diagnosis of familial hypercholesterolemia.Methods:Libraries were prepared for next-generation sequencing by two target enrichment protocols; one using the SureSelect Target Enrichment System and the other using the PCR-based Access Array platform.Results:In the validation cohort, both protocols showed 100% specificity, whereas the sensitivity for short variant detection was 100% for the SureSelect Target Enrichment and 98% for the Access Array protocol. Large deletions/duplications were only detected using the SureSelect Target Enrichment protocol. In the prospective cohort, the mutation detection rate using the Access Array was highest in patients with clinically definite familial hypercholesterolemia (67%), followed by patients with possible familial hypercholesterolemia (26%).Conclusion:We have shown the potential of target enrichment methods combined with next-generation sequencing for molecular diagnosis of familial hypercholesterolemia. Adopting these assays for patients with suspected familial hypercholesterolemia could improve cost-effectiveness and increase the overall number of patients with a molecular diagnosis.
Genotyping-by-Sequencing (GBS): A Novel, Efficient and Cost-Effective Genotyping Method for Cattle Using Next-Generation Sequencing.
High-throughput genotyping methods have increased the analytical power to study complex traits but high cost has remained a barrier for large scale use in animal improvement. We have adapted genotyping-by-sequencing (GBS) used in plants for genotyping 47 animals representing 7 taurine and indicine breeds of cattle from the US and Africa. Genomic DNA was digested with different enzymes, ligated to adapters containing one of 48 unique bar codes and sequenced by the Illumina HiSeq 2000. PstI was the best enzyme producing 1.4 million unique reads per animal and initially identifying a total of 63,697 SNPs. After removal of SNPs with call rates of less than 70%, 51,414 SNPs were detected throughout all autosomes with an average distance of 48.1 kb, and 1,143 SNPs on the X chromosome at an average distance of 130.3 kb, as well as 191 on unmapped contigs. If we consider only the SNPs with call rates of 90% and over, we identified 39,751 on autosomes, 850 on the X chromosome and 124 on unmapped contigs. Of these SNPs, 28,843 were not tightly linked to other SNPs. Average marker density per autosome was highly correlated with chromosome size (coefficient of correlation = -0.798, r(2) = 0.637) with higher density in smaller chromosomes. Average SNP call rate was 86.5% for all loci, with 53.0% of the loci having call rates >90% and the average minor allele frequency being 0.212. Average observed heterozygosity ranged from 0.046-0.294 among individuals, and from 0.064-0.197 among breeds, with Brangus showing the highest diversity as expected. GBS technique is novel, flexible, sufficiently high-throughput, and capable of providing acceptable marker density for genomic selection or genome-wide association studies at roughly one third of the cost of currently available genotyping technologies.
Targeted sequencing of cancer-related genes in colorectal cancer using next-generation sequencing.
Recent advance in sequencing technology has enabled comprehensive profiling of genetic alterations in cancer. We have established a targeted sequencing platform using next-generation sequencing (NGS) technology for clinical use, which can provide mutation and copy number variation data. NGS was performed with paired-end library enriched with exons of 183 cancer-related genes.

来源:生物探索
以下就是麦肯锡列举的颠覆性技术及其潜在的经济影响程度(含消费者盈余在内,即消费者并未支付的因创新而获得的价值),当然,这种影响评估只是粗略的,不会像 GDP 数字那么具体。

到 2025 年的影响力:
经济:0.7—1.6 万亿美元
生活:通过快速疾病诊断、新药物等延长及改善 75% 的生命
主要技术包括:先进 DNA 序列技术 ;DNA 综合技术;大数据及先进分析。
下一代基因组学中,尤为重要的是下一代测序技术、基因诊断以及基于遗传数据的个体化医疗。其中,下一代测序技术将产生高通量遗传数据,研究人员将这些海量的遗传数据与疾病进行关联(GWAS分析),从而提供疾病诊断和个体化医疗的靶向分子。
下一代基因组测序技术的3大产品
下一代基因组测序以单分子实时为特点,在纳米孔介质的基础上通过记录PH值、荧光和电流的改变,进而测出特定片段的碱基序列。目前,上市和待上市的下一代测序仪从技术特点上分为3个,它们是Life Technologies公司的Ion Torrent、Pacific Bioscience公司的PacBio RS和Oxford Nanopore Technologies公司的纳米孔测序仪。
Life Technologies公司的Ion Torrent
其核心技术是使用半导体技术在化学和数字信息之间建立直接的联系。在半导体芯片的微孔中固定DNA链,随后依次掺入ACGT。随着每个碱基的掺入,释放出氢离子,在它们穿过每个孔底部时能被检测到,通过对H+ 的检测,实时判读碱基。
Ion Torrent 半导体测序芯片的每个微孔里微球表面含有大约100 万个DNA 分子拷贝。测序时一个个核苷酸分子连续流过芯片微孔。如果核苷酸与特定微孔中的DNA 分子互补,则该核苷酸被合成到DNA 分子中,并且释放氢离子,该孔溶液的PH 发生变化。离子传感器检测到PH 变化后,即刻便从化学信息转变为数字电子信息。如果DNA 链含有两个相同的碱基,则记录电压信号是双倍的。
如果碱基不匹配,则无氢离子释放,也就没有电压信号的变化。这种方法属于直接检测DNA 的合成,因少了CCD 扫描,荧光激发等环节,几秒钟就可检测合成插入的碱基,大大缩短了运行时间。
Pacific Bioscience公司的PacBio RS
该测序仪的工作原理是,将基因组的DNA断开成许多很小的片段,这些小DNA片段制成溶液后滴入测序仪内的金属薄片上,金属薄片中分布着3000个纳米级小孔,这些纳米孔的直径不到70纳米,被滴入薄片上后,小DNA片段会分散到不同的纳米孔中。每个纳米孔中涂有一种特殊的酶——DNA聚合酶,DNA聚合酶 的特性是,能够沿着DNA片段的双链结构游动,在游动的过程中将DNA片段的双链结构打开,分成两个片段,也可为某个DNA单链找到对应的片段,重新组合 在一起,科学家将DNA聚合酶的作用形象地比喻成衣服的拉链,可开可合。
测序仪就是利用DNA聚合酶来“窃取人类的自然本性”的。一旦 DNA片段在纳米孔中分散完毕,向聚合酶分子滴入经过特殊处理的核苷酸溶液,谜底就会被揭开。核苷酸溶液中的每个核苷酸都用磷光染色剂做过标记,金属薄片 的底部也有一个缩微版光谱仪,一旦DNA片段中有核苷酸与之配对,标记物就会发出特定颜色的光,缩微版光谱仪就能检测并记录下这些闪光,测序仪从而记录下 每个DNA片段中的碱基对顺序。当闪光记录完之后,结果会输入计算机中,计算机破译出所有的DNA片段结果,并重新“组装”,让基因组恢复原样。
Oxford Nanopore Technologies公司的纳米孔测序仪
该测序仪有望在2013年上市,其工作原理是,单条DNA链将穿过蛋白孔,而第二种蛋白质将控制穿过的速度。随着碱基穿过纳米孔,它们改变了电流,带来特定的碱基组合。在理想的情况下,人们会看到每个碱基的典型模式,但目前的仪器将产生碱基三联体特有的模式,还需要通过计算机去卷积。
Oxford Nanopore Technologies公司在2012基因组生物学技术进展年会(AGBT)上宣布推出第一款商业化的纳米孔测序仪,引起了科学界的极大关注。纳米孔测序的读长很长,大约在几十kb,甚至100 kb;错误率目前介于1%至4%,且是随机错误,而不是聚集在读取的两端;数据可实时读取;通量很高(GridION有望在一天内以30倍覆盖度测序人类基因组);起始DNA在测序过程中不被破坏;以及样品制备简单又便宜。
具有突破意义的基因诊断
在最近召开的美国癌症研究学会(AACR)上,Mizuho Securities机构的分析师指出,基因诊断明显是肿瘤学的重要组成部分,即便大多数与会人士认为短期内分子诊断不能取代现存的诊断法,但是肿瘤基因组测序这一领域仍让多数人持乐观态度。报告提到了,更好的生物标记分子和诊断法被迫切需要。
研究人员和临床医生利用测序技术,以及基因组学、蛋白质组学和代谢组学方法去识别生物标记分子,促使其应用于遗传诊断、药物研发和临床药物测试的患者分类。
目前在分子诊断领域,最吸引眼球的新闻莫过于好莱坞女星兼导演安吉丽娜•朱莉因携带PCA缺陷基因而切除乳腺。她向《纽约时报》透露,因携带了母亲(因癌症去世)的癌症高危基因,所以不得不接受双乳乳腺切除预防乳癌手术治疗。这一PCA1缺陷基因让她患上乳癌几率高达87%,患卵巢癌几率50%。因此,朱莉2月主动选择做双乳乳腺切除降低患癌风险,手术已经在4月27日全部完成。
这一事件让“个人基因诊断”成为热门词汇。复旦大学生命科学院遗传学研究所副教授朱焕章接受采访时称:“基因检测的科学性是确定的,这也是未来的一个发展方向,将主要往个体化治疗、个体化诊断方向发展。每个人的情况都不一样,根据基因检测出个体的问题,就能有针对性地从根本上找出解决方案。”
个体化医疗前景值得期待
基因组时代已经到来,从基因组学角度实现传统医学的转化,通过分析基因组便可根据遗传信息掌握以前被称为“体质”的、由基因决定的个体特点,从而让个体化医学用于疾病预防和治疗的不同阶段。
美国贝勒人类基因组测序中心和有关机构联合发布一份报告称,贝勒大学医学院的研究人员及专家通过对一对双胞胎和他们的哥哥、父母的全基因组测序并进行对比分析,找到了引起这对双胞胎患有多巴反应性肌张力障碍疾病的突变基因,从而使医生能够对他们的病症进行针对性的治疗,并取得了良好的效果。贝勒人类基因组测序中心主任Richard Gibbs博士说:“我们有能力利用全基因组测序造福患者,这是一个巨大的进步。”
http://v.youku.com/v_show/id_XMjc2MjgwNzM2.html
个体化治疗患有多巴反应性肌张力障碍的双胞胎
另一个将基因组测序应用于个体化医疗的幸运儿是,患有白血病的华盛顿大学研究癌症的博士卢卡斯•沃特曼罹,在他病情恶化时,华盛顿大学里26台基因测序仪器中的一台,以及一台超级计算机,为沃特曼昼夜不停地运行。奇迹真的发生了!研究者们找到了元凶:一个正常基因FLT3表达 过于活跃,刺激了沃特曼癌细胞的快速生长增殖。更令人欣慰的是,他们找到了一种可能抑制问题基因表达的新药,这种新药之前只被批准用于治疗晚期肾癌。沃特曼成了第一个用这种药治疗白血病的病人。
基因组测序在个体化医疗成功运用的案例还是少数,已故的苹果公司前掌门人乔布斯当初对胰腺癌束手无策时,就曾联系医生进行过基因组测序和分析,不惜花费10万美元希望通过基因组测序找到胰岛细胞瘤的致病基因,从而实现个体化医疗。据乔布斯传记的作者艾萨克森透露,乔布斯本人也曾对此抱以巨大期望,他称自己要么是最早击败癌症的人之一,要么属于最后一批死于癌症的人。
文献链接:
The use of next-generation sequencing in clinical diagnosis of familial hypercholesterolemia.
Purpose:Familial hypercholesterolemia is a common Mendelian disorder associated with early-onset coronary heart disease that can be treated by cholesterol-lowering drugs. The majority of cases in the United Kingdom are currently without a molecular diagnosis, which is partly due to the cost and time associated with standard screening techniques. The main purpose of this study was to test the sensitivity and specificity of two next-generation sequencing protocols for genetic diagnosis of familial hypercholesterolemia.Methods:Libraries were prepared for next-generation sequencing by two target enrichment protocols; one using the SureSelect Target Enrichment System and the other using the PCR-based Access Array platform.Results:In the validation cohort, both protocols showed 100% specificity, whereas the sensitivity for short variant detection was 100% for the SureSelect Target Enrichment and 98% for the Access Array protocol. Large deletions/duplications were only detected using the SureSelect Target Enrichment protocol. In the prospective cohort, the mutation detection rate using the Access Array was highest in patients with clinically definite familial hypercholesterolemia (67%), followed by patients with possible familial hypercholesterolemia (26%).Conclusion:We have shown the potential of target enrichment methods combined with next-generation sequencing for molecular diagnosis of familial hypercholesterolemia. Adopting these assays for patients with suspected familial hypercholesterolemia could improve cost-effectiveness and increase the overall number of patients with a molecular diagnosis.
Genotyping-by-Sequencing (GBS): A Novel, Efficient and Cost-Effective Genotyping Method for Cattle Using Next-Generation Sequencing.
High-throughput genotyping methods have increased the analytical power to study complex traits but high cost has remained a barrier for large scale use in animal improvement. We have adapted genotyping-by-sequencing (GBS) used in plants for genotyping 47 animals representing 7 taurine and indicine breeds of cattle from the US and Africa. Genomic DNA was digested with different enzymes, ligated to adapters containing one of 48 unique bar codes and sequenced by the Illumina HiSeq 2000. PstI was the best enzyme producing 1.4 million unique reads per animal and initially identifying a total of 63,697 SNPs. After removal of SNPs with call rates of less than 70%, 51,414 SNPs were detected throughout all autosomes with an average distance of 48.1 kb, and 1,143 SNPs on the X chromosome at an average distance of 130.3 kb, as well as 191 on unmapped contigs. If we consider only the SNPs with call rates of 90% and over, we identified 39,751 on autosomes, 850 on the X chromosome and 124 on unmapped contigs. Of these SNPs, 28,843 were not tightly linked to other SNPs. Average marker density per autosome was highly correlated with chromosome size (coefficient of correlation = -0.798, r(2) = 0.637) with higher density in smaller chromosomes. Average SNP call rate was 86.5% for all loci, with 53.0% of the loci having call rates >90% and the average minor allele frequency being 0.212. Average observed heterozygosity ranged from 0.046-0.294 among individuals, and from 0.064-0.197 among breeds, with Brangus showing the highest diversity as expected. GBS technique is novel, flexible, sufficiently high-throughput, and capable of providing acceptable marker density for genomic selection or genome-wide association studies at roughly one third of the cost of currently available genotyping technologies.
Targeted sequencing of cancer-related genes in colorectal cancer using next-generation sequencing.
Recent advance in sequencing technology has enabled comprehensive profiling of genetic alterations in cancer. We have established a targeted sequencing platform using next-generation sequencing (NGS) technology for clinical use, which can provide mutation and copy number variation data. NGS was performed with paired-end library enriched with exons of 183 cancer-related genes.

来源:生物探索
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发