如此研究疾病,高分文章还远么?
| 导读 | 2015年,美国国立卫生研究院(NIH)宣布,将人类染色体的三维结构研究方向视为进一步认知疾病的重大机遇,并批准立项为共同基金重大项目,即“3D 核小体”(3D-Nucleome)计划,至此开启了基因组学和生物学的后测序时代。 |

2015年,美国国立卫生研究院(NIH)宣布,将人类染色体的三维结构研究方向视为进一步认知疾病的重大机遇,并批准立项为共同基金重大项目,即“3D 核小体”(3D-Nucleome)计划,至此开启了基因组学和生物学的后测序时代。
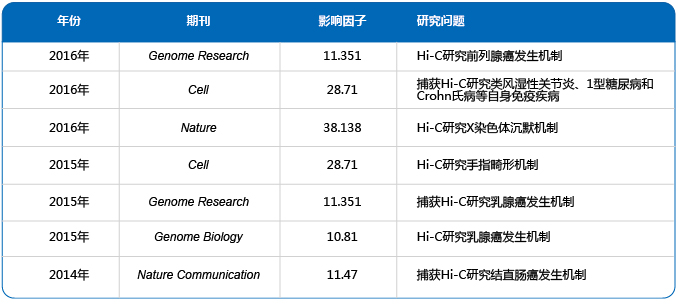
表 Hi-C在疾病中的文章发表情况
一、Hi-C研究乳腺癌发生机制
本篇文章发表在Genome Biology上(IF=10.81),选取人乳腺上皮细胞系MCF-10A和乳腺癌细胞系MCF-7,文章思路简单清晰,主要是在不同分辨率下进行染色体三维结构的比较,我们具体来瞄一瞄。
1. 样本选择:
本文选取两种细胞系即人乳腺上皮细胞系MCF-10A和乳腺癌细胞系MCF-7。
2. 文库及测序策略:
文库:Hi-C文库+转录组文库
测序策略: RNA-seq:HiSeq 2000 SE100
Hi-C:HiSeq 2000 PE100
3. 研究思路
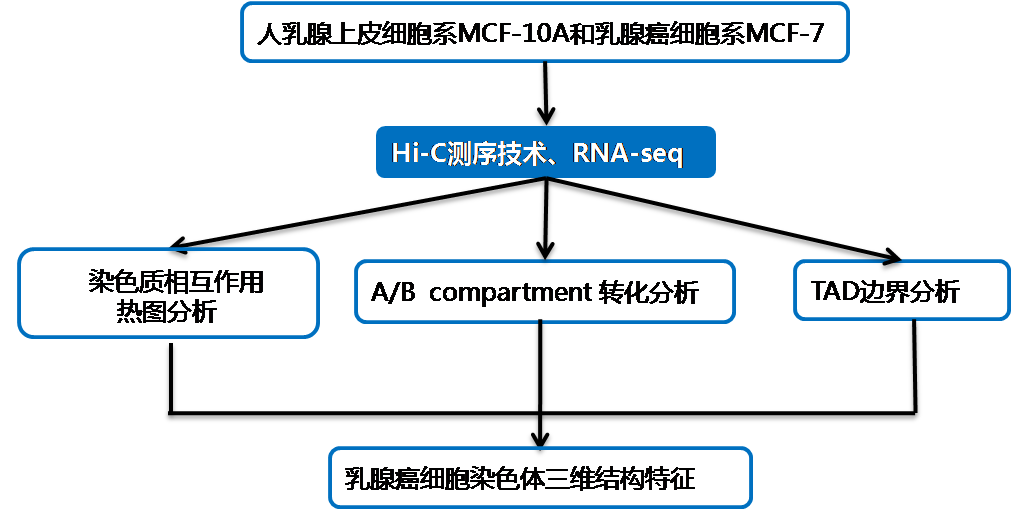
图1 文章研究思路
(1)1M分辨率下人乳腺上皮细胞系MCF-10A和乳腺癌细胞系MCF-7在染色体互作上的差异
通过染色体相互作用热图比较,肉眼是很难寻找二者的差异,或者说观察到的差异不明显,本文采取“减法”算法,巧妙地将两个细胞系的互作热图整合到一张图中,结果发现在小染色体中(16-22号染色体),MCF-10A细胞系中有较强的基因相互作用。
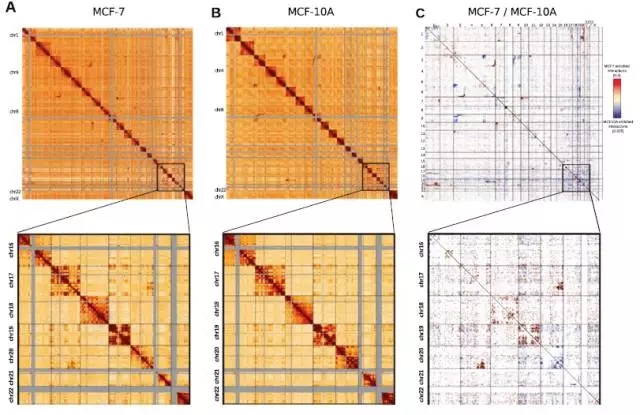
图2 MCF-10A和MCF-7中16-22号染色体间相互作用比较
之前的研究表明,基因组内的相互作用有两种模式:开放区((A-type)和封闭区((B-type)。在乳腺癌细胞系MCF-7和乳腺上皮细胞系MCF-10A基因组中鉴定出了这两种区室,MCF-10A、MCF-7的开放和封闭区域分布大体上非常相似,只有一些区域有A到B区室或者B到A区室的转化。这些转化区域中的很多基因和癌症重要通路WNT相关。
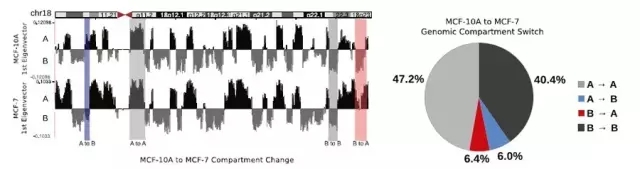
图3 A/B compartment 差异分析
MCF-10A和MCF-7基因组中大规模染色体相互作用差异和改变的基因区室内是否会对TAD形成和基因表达造成影响是未知的。为解决这个问题,用40kb分辨率来鉴定TAD边界, 发现一些TAD边界是乳腺癌细胞系特有的。
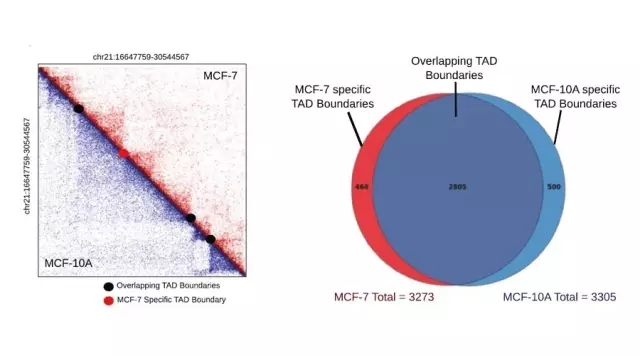
图4 TAD差异分析
二、捕获Hi-C研究类风湿性关节炎、1型糖尿病和Crohn氏病等自身免疫疾病
本篇文章是本月18号由英国Babraham研究所Peter Fraser博士研究团队发
表在cell上,Babraham研究所一直专注于捕获Hi-C方法的研发和应用,文章先后发表在《Genome Research》、《Nature Genetics》上。
1.样本选择:
17种不同类型的血细胞,含有Naive CD4+ T cells、Megakaryocytes、Neutrophils
等自身免疫疾病相关的细胞系。
2.文库及测序策略:
文库: Hi-C、Capture Hi-C
测序策略:Illumina HiSeq2500
3.研究结果:
通过对17种不同类型的血细胞中与基因相互作用的调控元件调查,本文建立起了在各个细胞类型中调节特定基因的远程调控元件的集合。远程调控元件区域的一些微小变化,会干扰基因的正常调控,导致特定疾病的发生。本研究发现了成千上万与特定疾病相关的新基因,包括类风湿性关节炎、1型糖尿病和Crohn氏病等疾病。
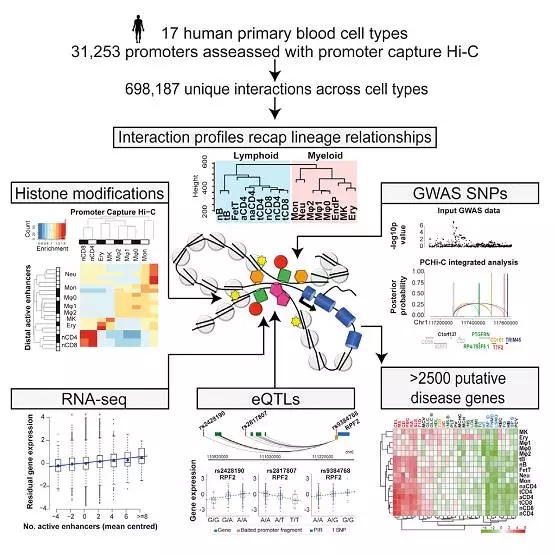
图5 捕获Hi-C揭示疾病相关机制
三、Hi-C揭示手指畸形机制
本篇文章发表在cell上,采用的测序技术种类以及样本丰富程度都是第一篇乳腺癌文章达不到的。小编带大家一起来详细地看看,NCS级别的文章如何设计的。
1. 样本选择:
手指畸形病人的血液样本、皮肤组织样本,野生小鼠胚胎干细胞、CRISPR编辑后的突变小鼠模型。
2. 测序技术种类:
Hi-C、4C-seq、人全基因组测序、外显子测序、RNA-seq、ChIP-seq等。
3. 研究思路:
本文从病人样本出发,采取人重测序寻找手指畸形对应的染色体变异,接着采取小鼠模型验证染色体变异是否引起染色体三维结构的变化,最终回归病人样本揭示手指畸形的机制。发现是染色体变异(缺失、复制、倒位)引起了染色体三维结构的变化,使原来的TAD边界被打破;从而远程调控元件与目的基因间的互作发生改变,引起基因表达水平改变,造成手指畸形。
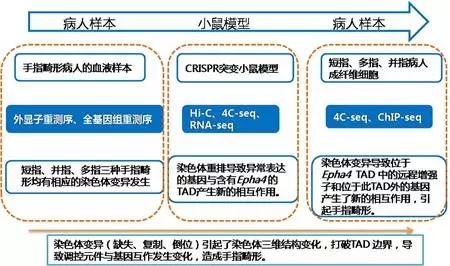
图6 研究思路
4. 结果展示:
(1)人重测序寻找手指畸形相关的染色体变异
对手指畸形病人血液样本进行人全基因组重测序,发现在三种手指畸形中均有相应的染色体变异发生。 短指:染色体缺失,包含EPHA4 及PAX3 非编码区的一端区域,并指:染色体倒位和区域复制,多指:染色体区域复制。

图7 手指畸形样本的染色体变异情况
(2)CRISPR 方法构建手指疾病的相关小鼠模型,探究染色体变异引起的染色体三维结构变化
在小鼠模型中用4C-seq发现染色体重排导致异常表达的基因与含有Epha4 的TAD产生新的相互作用,即染色体变异引起染色体三维结构发生变化(TAD的边界改变)。 此外,本文返回到病人样本,短指、多指、并指病人成纤维细胞4C-seq结果与小鼠手指4C-seq和人Hi-C结果高度相似,在病人细胞系中已破坏的TAD导致相互作用异常。
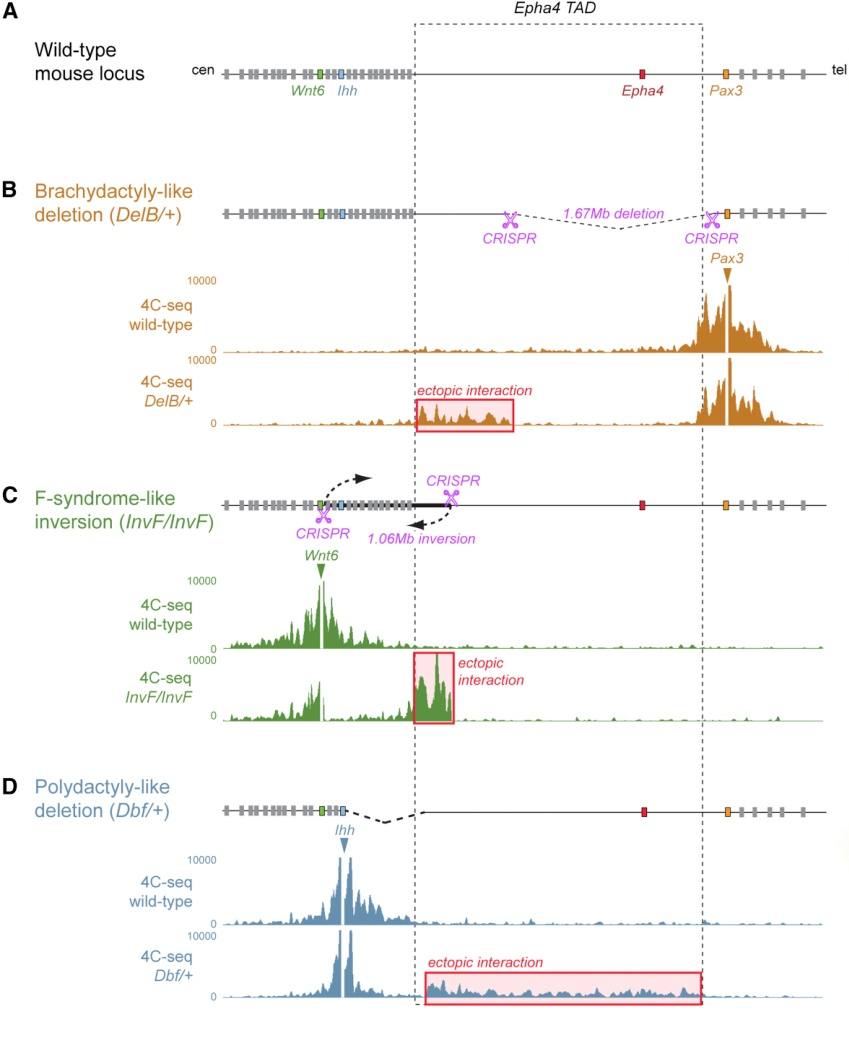
图8 染色体变异引起染色体三维结构变化
(3) TAD边界改变导致远程调控元件和目的基因相互作用发生变化
结合已有的ChIP-seq数据发现,边界发生变化的TAD中含有重要的增强子,这些增强子与相邻TAD内的基因有异常的互作,导致调控紊乱,造成手指畸形。

图9 增强子与基因互作紊乱
Hi-C作为安诺优达特色技术,不断探索不断创新?
可以揭示染色体三维结构,且可用于辅助组装
从群体细胞Hi-C、1kb Hi-C、捕获Hi-C
单细胞Hi-C拓展到Hi-C辅助组装
物种经验跨越动植物及微生物
合作单位贯穿国内外科研院所
安诺高端Hi-C诚邀您的加入...
参考文献:
[1]Taberlay P C, Achinger-kawecka J, Lun A T, et al. Three-dimensional disorgan isation of the cancer genome occurs coincident with long range genetic and epigenetic alterations.[J]. Genome Research, 2016.
[2]Javierre B M, Burren O S, Wilder S P, et al. Lineage-specific genome architecture links enhancers and non-coding disease variants to target gene promoters[J]. Cell.
[3]Giorgetti L, Lajoie B R, Carter A C, et al. Structural organization of the inactive X chromosome in the mouse[J]. Nature, 2016, 535(7613).
[4]Lupiá?ez D G, Kraft K, Heinrich V, et al. Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions[J]. Cell, 2015, 161(5):1012-25.
[5]Dryden N H, Broome L R, Dudbridge F, et al. Unbiased analysis of potential targets of breast cancer susceptibility loci by Capture Hi-C[J]. Genome Research, 2014, 24(11):1854-1868.
[6]Barutcu A R, Lajoie B R, Mccord R P, et al. Chromatin interaction analysis reveals changes in small chromosome and telomere clustering between epithelial and breast cancer cells[J]. Genome Biology, 2015, 16(1):1-14.
[7]J?ger R, Migliorini G, Henrion M, et al. Capture Hi-C identifies the chromatin interactome of colorectal cancer risk loci[J]. Nature Communications, 2015, 6.(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发