展望NGS的下一个突破点,RNA-Seq和线粒体测序上榜
| 导读 | 如今NGS已能够快速经济地阅读数亿个reads,所及之处远超越基因组学(如RNA测序使转录组学发生革命性变化)。尽管NGS取得了诸多进步,但依然存在一些重大的挑战。 |

如今NGS已能够快速经济地阅读数亿个reads,所及之处远超越基因组学(如RNA测序使转录组学发生革命性变化)。尽管NGS取得了诸多进步,但依然存在一些重大的挑战。日前,牛津大学举办的“NGS七周年大会”聚焦了NGS面临的挑战,此次会议阐述了NGS领域最令人生畏的障碍,同时也阐述了跨越这些障碍的技术。
本次会议中提及的NGS的新突破点包括毒性、RNA-Seq文库及可变剪接图谱中的多余转录组的去除以及线粒体DNA测序。如今NGS在线粒体DNA测序领域取得了一定的进展,线粒体在疾病发展中的作用逐渐被认识,然而这些细胞器中的DNA测序十分复杂,因为线粒体具有相当大的遗传复杂性和异质性。NGS的另一个突破点为肿瘤异质性分析,长期以来肿瘤分析都被异质性困扰着,但幸运的是,异质性肿瘤样本可通过排序程序将病变细胞从总群体中分离出来,且这些细胞群更适合测序。消除毒性转录组(Toxic Transcripts)
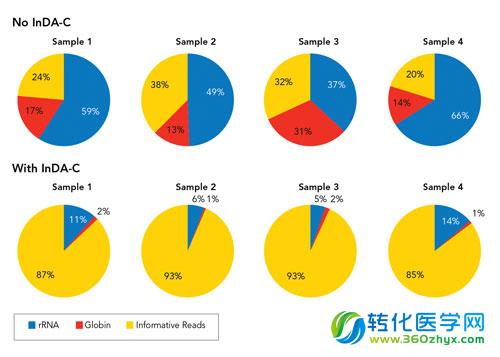
InDA-C技术效应图
创建高特性的RNA-Seq文库仍面临着长期的挑战,NuGEN Technologies技术总监 Luke Sherlin博士说,“减少不必要转录组如rRNA、球蛋白以及其他管家类的转录组,同时保持总RNA群中的必要转录组十分有必要。” NuGEN Technologies公司开发了新技术来实现这个目标,他们在RNA-Seq文库构建的过程中,利用InDA-C(Insert-Dependent Adaptor Cleavage )方法(使用特定的酶)来消除文库中多余的转录组序列,这种方法与杂交捕获方法形成对比。“InDA-C是一种灵活的方法,容易应用于各种不同的物种如人类、小鼠、大鼠、果蝇和拟南芥等”,Sherlin说,“InDA-C技术是一种相对较新的方法,可提供一种独特的方式来构造任何物种的无偏差的RNA-Seq文库,且定制步骤方便简单。”可变剪接和RNA-Seq数据库
RNA-Seq技术还提供了一个宝贵的工具来破译转录组的可变剪接。有些基因的一个mRNA前体通过不同的剪接方式(选择不同的剪接位点)产生不同的mRNA剪接异构体,这一过程称为可变剪接(或选择性剪接, alternative splicing) 。可变剪接是调节基因表达和产生蛋白质组多样性的重要机制, 是导致真核生物基因和蛋白质数量较大差异的重要原因。
霍普金斯大学医学院遗传医学研究所Liliana Florea教授表示,“人类基因组计划10年前创建了剪接变异初始图谱,但该图谱仍不完整,仍欠缺包含所有可变剪接的存储库。每个剪接变异体的编目带来的挑战是令人畏惧的,但RNA-Seq技术可深入分析细胞和组织的转录组,提供一种可详细描述可变剪接的技术手段。但RNA-Seq仍然很难准确拼凑长异构体所产生的小片段。
Florea教授报告称,她和她的同事们决定开发适用于RNA-Seq数据的计算机系统,“我们决定开发其他程序无法达到的算法来确定二次变异。”使用这种工具,她希望能够明确可变剪接是如何发生以及如何随细胞类型发生变化。该研究团队曾利用Illumina公司的Body Map dataset建立每个组织的可变剪接的综合目录,该目录囊括了16个组织的25亿个序列,被用于不同组织的比较。然而这种比较是一个艰巨的任务,为了保持比较的精度,研究人员依赖于内部软件套件SpliceBox,该研究小组发现,超过60%的发现是新的,包括新的外显子、新的内含子等。
“这种方法可对现有的变异注释数据库进行补充,还可为可变剪辑的进化和发展提供新的见解”, Florea教授强调,“但更多的RNA-Seq分析需要进一步评估,我们希望更多的实际应用,包括临床测序、基本分子生物学、癌症研究、甚至包括植物基因组学。”
线粒体DNA测序
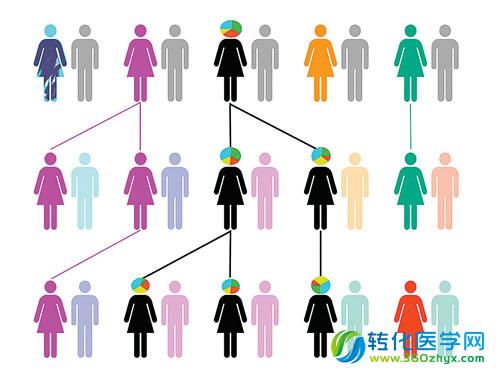
异质性混合物从母亲遗传给后代
线粒体,这些神奇的细胞体,不仅产生ATP供应能量,还与人体罹患癌症、心脏病和糖尿病的风险有关。西奈山医院肿瘤学教授Ravi Sachidanadam博士引领的研究证实了这些观点。Sachidanadam教授表示,“真核细胞有两个基因组——核DNA(nDNA)和线粒体DNA(mtDNA),虽然线粒体的活性取决于上千个蛋白质,这些蛋白主要由核DNA编码,但由线粒体DNA编码的蛋白(大约13个以上)也扮演了关键的角色。”线粒体DNA测序不是一件小事,因为每个线粒体携带了多个线粒体基因组(5-10个),且每个细胞拥有成百上千的线粒体。Sachidanadam博士承认,准确编目线粒体DNA多样性是一项挑战,虽然线粒体DNA丰度不及总DNA的1%,但它在细胞之间多重复合,同时核DNA测序结果经常被线粒体DNA混淆。
为了克服这些挑战,Sachidanadam博士及其同事开发了一种称为MSeek的线粒体测序技术。Sachidanadam博士说,“将线粒体孤立起来相当苦难,我们的方法包括通过耗尽线性核DNA及廉价测序来净化线粒体DNA,MSeek可产生高纯度的线粒体DNA(>90%),所达的灵敏度和特异性前所未有,这个方法是净化和检测线粒体DNA的新方法。”
该研究团队所取的重大突破是确定了核酸外切酶V是消化核DNA的最佳酶,该酶同时可保持线粒体DNA圆形完好无损。Sachidanadam博士补充说,他们利用Illumina公司的Miseq测序平台来展开这个试验,并计算假基因的含量。
使用MSeek技术,该研究团队得到了一个惊人的发现,“我们不仅证实了异质性无处不在(多个线粒体DNA单倍体的存在),我们还发现了异质性可作为细胞类型的识别指纹,此外我们还发现细胞之间可互相交换线粒体DNA,这影响了单个细胞中线粒体DNA的稳定性。”
Sachidanadam博士说,“我们团队现研究成果仅仅是MSeek技术所揭露的一小部分,虽然MSeek技术目前面临的局限为所需的DNA总量至少为4ug,我们预计该技术将被应用到其他领域中,不仅包括治疗,还包括取证等。
从FFPE中分离出纯的肿瘤细胞
根据Silicon Biosystems首席商务官Raimo Tanzi博士,肿瘤分析通常因为异质性而存在困扰,数字分析方法的引进,如NGS和数字PCR可助于梳理少数的肿瘤样本,然而只有均质抽样才能提供最清晰的解释。为了解决这些问题,Silicon Biosystems应用了新的数码技术(DEPArray?)从异构的肿瘤样本中分离出纯细胞群,该半导体技术基于在CMOS芯片上利用双向电泳(DEP)将单细胞与单级电泳结合。
Tanzi博士说,“FFPE样本最初先被转化为细胞悬浊液,并被给予适当的标记,之后置于一次性的微流控盒中,一旦处于流动细胞状态,每个细胞先被CMOS芯片控制电极捕获,然后被显微镜扫描并获得荧光图像。根据这些图像,每个细胞被识别为特定的类型(肿瘤、基质等),最终同种类型的细胞纯度在100%。”
新技术可将几十到几百个纯细胞从细胞池中分离出来并用于全基因组分析。细胞池中可能包括上皮-间充质转化细胞、肿瘤浸润淋巴细胞和间质细胞。Tanzi博士说,“DEPArray是第一个自动化技术可从异构的FFPE样本中分离出纯的细胞,这给许多研究带来了好处,其中包括一些与癌症相关的研究,如样品的均匀性、明确识别拷贝变异数、杂合子丢失等。该技术的另一个可能用途是进行非常小的样本的遗传分析,包括细针穿刺标本和肿瘤低细胞结构的遗传分析。”显然,研究人员在NGS领域取得了令人兴奋的进展,未来应该有更快、更精确、更新颖的技术。
提高文库制备的性能
文库制备是NGS的重要部分,成功的测序需要高质量的文库来产生足够的收益。由于测序技术的提高以及范围的扩大都受文库建设的推动。高性能的分析是十分必要的,包括低输入量以及低质量样本或包含极端CG含量样本的分析。同时需要协议来保证文库的质量。新英格兰生物实验室研究人员表示他们最近重新研制了NEBNNextUltra DNA工作流程中的试剂配方,为Illumina创建 NEBNext Ultra II DNA文库制备试剂盒(NEBNext Ultra II DNA Library Prep Kit)。根据NEBNext开发组组长Eileen Dimalanta博士,新的试剂能使客户克服了文库制备面临的挑战,如复杂的样品类型,FFPEDNA、样本中CG含量的统一以及PCR循环数等。(转化医学网360zhyx.com)
 腾讯登录
腾讯登录
还没有人评论,赶快抢个沙发