【快讯】浅谈|分子诊断技术分类
| 导读 | 分子诊断技术的分类 |
从技术上看,分子诊断领域主要包括PCR(传统PCR、qPCR和dPCR)、荧光原位杂交(FISH)、基因芯片和第二代高通量测序技术(NGS)等。其中,PCR是目前应用最成熟、市场份额最大的技术平台,在国内分子诊断中市占率高达40%;其次是分子杂交技术,占比为35%。
1. 多聚酶链式反应(Polymerase Chain Reaction,PCR)
上世纪八十年代,Cetus Corporation公司的化学家Kary Mullis发明了PCR,对分子生物学乃至整个生命科学界产生了巨大的推动作用。从此以后,基于PCR的DNA扩增技术逐渐成为生物学研究的基础,开启了一个又一个生命科学领域的重大课题。目前,PCR在传染性疾病(细菌、真菌、病毒、支原体)诊断、NIPT、干细胞研究、生物标志物开发等方面有着极为广泛的应用。
PCR是指在DNA多聚酶(又称“DNA聚合酶”,如Taq DNA聚合酶)催化条件下,以母链DNA为模板,以特定引物为延伸起点,通过高温变性(94℃)、低温复性(55℃)、中温延伸(72℃)等步骤,在体外复制出与母链模板DNA互补的子链DNA的过程。PCR是生物体外的特殊DNA复制,最大的特点是能将微量的DNA大幅扩增。
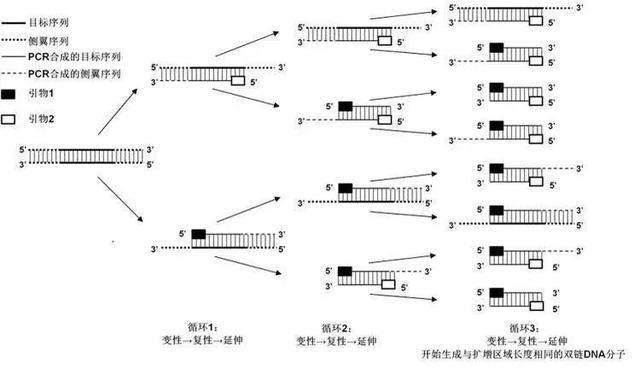
图:PCR原理
与杂交技术、高通量测序技术相比,PCR技术主要优势在于高灵敏度、易于推广,主要局限在于检测位点单一且已知,多重基因联合检测时可涵盖的基因数量受限。但从短期来看,PCR技术仍将是分子诊断主流技术平台。
PCR技术自问世以来,经历了三十余年的高速发展,目前已经成为世界上应用最成熟、市场份额最大的分子诊断技术平台。研究者们在该技术原理基础之上开发出一系列衍生的分子检测技术,主要可以分为三类:第一代PCR电泳法、第二代荧光定量PCR(qPCR)和第三代数字PCR(dPCR)。
1.1 PCR电泳法(传统PCR)
PCR电泳法是最原始、最简单的基于凝胶的低通量PCR方法,是指在PCR反应结束之后,通过琼脂糖凝胶电泳或毛细管电泳等方法,利用长波式紫外灯照射或放射自显影技术对PCR扩增产物进行定性检测分析的DNA检测方法。但PCR电泳法本身只能做定性检测而无法定量,因为该反应产出的DNA量不一定能反映最初情况,例如不同样品和序列的扩增效率是有差异的。此外,还存在交叉污染、检测耗时长等缺点,目前处于衰退期,基本上已被淘汰。
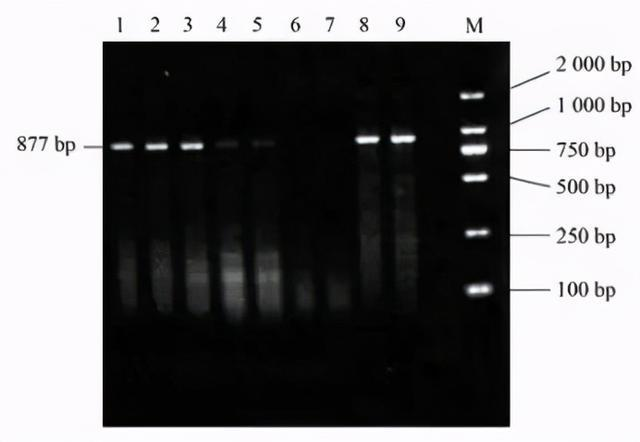
图:PCR电泳条带图
1.2 qPCR电泳法(荧光定量PCR)
qPCR是利用插入性染料或荧光探针对PCR产物进行标记跟踪,通过监控整个PCR过程中荧光信号的强度变化得到待测DNA样本初始浓度的相对定量分析方法。该方法相较于传统PCR方法而言,无需取出PCR产物进行分离,且可作定量分析,并具有特异性强、灵敏度高、自动化程度高、无污染等优势,目前已逐渐取代常规PCR,成为应用最成熟、临床应用最广泛的分子诊断技术平台。
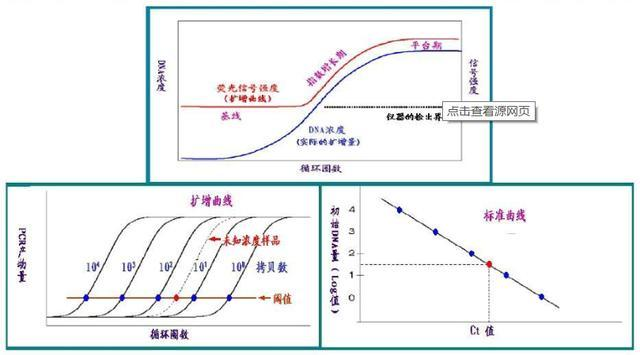
图:PCR标准曲线定量分析待测DNA样本浓度
根据引入荧光基团的不同,qPCR可分为荧光探针法定量PCR和荧光染料法定量PCR。
荧光探针法定量PCR的原理为:在PCR反应体系中,加入一对引物的同时加入一个特异性的荧光探针,该探针为一寡核苷酸,两端分别标记一个报告荧光基团和一个淬灭荧光基团。开始时,探针完整地结合在DNA任意一条单链上,报告基团发射的荧光信号被淬灭基团吸收,检测不到荧光信号;PCR扩增时,Taq酶将探针酶切降解,使报告荧光基团和淬灭荧光基团分离,从而荧光监测系统可接收到荧光信号。每扩增一条DNA链,就有一个荧光分子形成,实现了荧光信号的累积与PCR产物形成完全同步,这就是定量的基础所在。
荧光染料法定量PCR的原理为:在PCR反应体系中,加入过量荧光染料,该染料只与双链DNA小沟结合并发射荧光信号,并不与单链DNA链结合,而且在游离状态不发出荧光,从而保证荧光信号的增加与PCR产物的增加完全同步,此即定量的基础所在。
qPCR在国内外均为分子诊断临床应用最广泛的技术平台,尤其是在感染性疾病(HBV、STD等)和肿瘤伴随诊断领域,目前仍以qPCR技术平台为主。据不完全统计,截至2020年5月11日,国家药监局(NMPA)共批准了806个PCR类产品,其中荧光定量qPCR产品占比高达85.11%。在伴随诊断领域,NMPA获批的伴随诊断产品中有60%都是基于qPCR技术,而FDA批准的39个伴随诊断产品基于qPCR技术的产品占比也高达38.46%(15个)
1.3 dPCR(数字PCR)
dPCR是新兴起来的一种核酸分子绝对定量技术,主要采用当前分析化学热门研究领域的微滴化方法,对待测样本进行高倍稀释,分配到不同的反应单元内,每个单元或不含有、或含有一至多个目标分子(DNA模板),再将所有反应单元内的样本进行PCR扩增,最后结合泊松分布原理,分析软件根据阳性分子个数与比例得出靶分子的起始拷贝数(copies)或浓度(copies/uL)。与qPCR相比,dPCR具有绝对定量、高灵敏度、可耐受PCR反应抑制物、减弱背景序列干扰信号等显著优势。
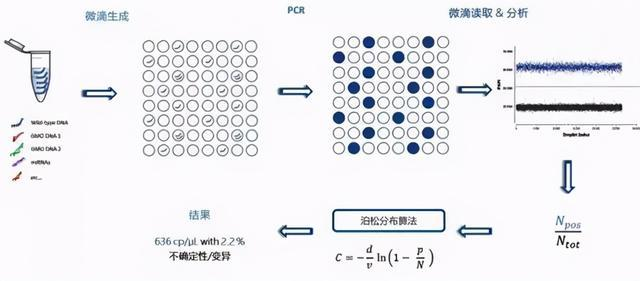
图:微滴式dPCR原理
dPCR在国内尚处于市场导入期,目前仅有南京科维思生物的HER2基因扩增检测试剂盒(数字PCR法)获批,在肿瘤伴随诊断、肿瘤早筛、传染病检测、NIPT、药物基因组学等领域具有较大临床应用潜力和优势。根据沙利文数据显示,中国dPCR行业市场规模从2013年的14.6亿元增加至2017年的72亿元,CAGR达到29.2%,到2022年市场规模预计将达到266.6亿元。
2 荧光原位杂交(Fluorescence In-Situ Hybridization,FISH)
FISH是一种按照核酸碱基互补配对原则,利用荧光素标记的、已知序列的DNA探针直接原位杂交到待测DNA样本模板上,经荧光检测系统和图形分析技术对待测DNA序列进行定位、定性和相对定量的技术。FISH主要用于指导肿瘤靶向药物使用、肿瘤预后、肿瘤疾病分型诊断、NIPT等领域,是检测HER-2基因(一种原癌基因)状态的金标准,目前在国内发展较为成熟,且国内大多数省份和地区均已将其纳入医保。
FISH操作流程为:首先用荧光素(直接标记法)或“生物素-荧光素标记亲和素”放大系统(间接标记法)标记探针核苷酸,利用切口平移法或随机引物法制备出带有荧光标记的、已知序列的DNA探针。在高温条件下使该探针与待测DNA样本模板发生变性并打开双链,令两者混合杂交并形成杂交体,最后通过荧光显微镜进行观察,从而实现对目标DNA的定性、定位和相对定量分析。
3 基因芯片技术
基因芯片技术又称DNA微阵列技术,是将大量荧光或酶标记的、序列已知的DNA探针集成在同一芯片上与样品分子进行杂交,通过采集荧光或化学发光信号获得样本序列信息。基因芯片技术可看作是FISH与芯片技术的结合,可以实现对大量目标基因同时进行检测,具有成本相对较低、检测效率较高的优势,主要应用于消费级基因检测、病毒分型、耐药突变位点检测、遗传基因和肿瘤基因检测等领域。
NGS又称为下一代测序技术,是指通过模板DNA分子的化学修饰,将其锚定在纳米孔或微载体芯片上,利用碱基互补配对原理,在DNA聚合酶链反应或DNA连接酶反应过程中,通过采集荧光标记信号或化学反应信号,实现碱基序列的解读。NGS最显著特征是高通量,一次性可完成几十万至上百万条序列的测定,使得对一个物种的转录组测序或基因组深度测序变得方便易行。该技术不需要荧光标记的核酸探针,也不需要进行电泳,具有分析结果快速、准确、灵敏度高和自动化的特点。
罗氏454生命科学公司于2007年推出的GS FLX测序系统,是一种基于焦磷酸测序原理而建立起来的高通量基因组测序系统,开创了第二代测序技术的先河。
随着NGS技术进步和测序成本的降低、肿瘤医生和病人对NGS检测认知不断完善、测序服务对象和应用细分领域的拓展、企业间合作的增加,NGS有望迎来快速发展。根据美国 Markets and Markets报告显示,预计全球高通量基因测序市场规模将从2019年的78亿美元增加至2025年的244亿美元,CAGR为20.9%。
 腾讯登录
腾讯登录






还没有人评论,赶快抢个沙发